Один из механизмов Active Directory (AD), с которым могут быть связаны всевозможные затруднения, это репликация. Репликация – критически важный процесс в работе одного или более доменов или контроллеров домена (DC), и не важно, находятся они на одном сайте или на разных. Неполадки с репликацией могут привести к проблемам с аутентификацией и доступом к сетевым ресурсам. Обновления объектов AD реплицируются на контроллеры домена, чтобы все разделы были синхронизированы. В крупных компаниях использование большого количества доменов и сайтов – обычное дело. Репликация должна происходить внутри локального сайта, так же как дополнительные сайты должны сохранять данные домена и леса между всеми DC.
В этой статье речь пойдет о методах выявления проблем с репликацией в AD. Кроме того, я покажу, как находить и устранять неисправности и работать с четырьмя наиболее распространенными ошибками репликации AD:
- Error 2146893022 (главное конечное имя неверно);
- Error 1908 (не удалось найти контроллер домена);
- Error 8606 (недостаточно атрибутов для создания объекта);
- Error 8453 (доступ к репликации отвергнут).
Вы также узнаете, как анализировать метаданные репликации с помощью таких инструментов, как AD Replication Status Tool, встроенная утилита командной строки RepAdmin.exe и Windows PowerShell.
Для всестороннего рассмотрения я буду использовать лес Contoso, который показан на рисунке. В таблице 1 перечислены роли, IP-адреса и настройки DNS-клиента для компьютеров данного леса.
 |
| Рисунок. Архитектура леса |
Для обнаружения неполадок с репликацией AD запустите AD Replication Status Tool на рабочей станции администратора в корневом домене леса. Например, вы открываете этот инструмент из системы Win8Client, а затем нажимаете кнопку Refresh Replication Status для уверенности в четкой коммуникации со всеми контроллерами домена. В таблице Discovery Missing Domain Controllers на странице Configuration/Scope Settings инструмента можно увидеть два недостающих контроллера домена, как показано на экране 1.
 |
| Экран 1. Два недостающих контроллера домена |
В таблице Replication Status Collection Details вы можете проследить статус репликации контроллеров домена, которые никуда не пропадали, как показано на экране 2.
 |
| Экран 2. Статус репликации контроллеров домена |
Пройдя на страницу Replication Status Viewer, вы обнаружите некоторые ошибки в репликации. На экране 3 видно, что возникает немалое число ошибок репликации, возникающих в лесу Contoso. Из пяти контроллеров домена два не могут видеть другие DC, а это означает, что репликация не будет происходить на контроллерах домена, которые не видны. Таким образом, пользователи, подключающиеся к дочерним DC, не будут иметь доступ к самой последней информации, что может привести к проблемам.
 |
| Экран 3. Ошибки репликации, возникающие в лесу Contoso |
Поскольку ошибки репликации все же возникают, полезно задействовать утилиту командной строки RepAdmin.exe, которая помогает получить отчет о состоянии репликации по всему лесу. Чтобы создать файл, запустите следующую команду из Cmd.exe:
Repadmin /showrel * /csv > ShowRepl.csv
Проблема с двумя DC осталась, соответственно вы увидите два вхождения LDAP error 81 (Server Down) Win32 Err 58 на экране, когда будет выполняться команда. Мы разберемся с этими ошибками чуть позже. А теперь откройте ShowRepl.csv в Excel и выполните следующие шаги:
- Из меню Home щелкните Format as table и выберите один из стилей.
- Удерживая нажатой клавишу Ctrl, щелкните столбцы A (Showrepl_COLUMNS) и G (Transport Type). Правой кнопкой мыши щелкните в этих столбцах и выберите Hide.
- Уменьшите ширину остальных столбцов так, чтобы был виден столбец K (Last Failure Status).
- Для столбца I (Last Failure Time) нажмите стрелку вниз и отмените выбор 0.
- Посмотрите на дату в столбце J (Last Success Time). Это последнее время успешной репликации.
- Посмотрите на ошибки в столбце K (Last Failure Status). Вы увидите те же ошибки, что и в AD Replication Status Tool.
Таким же образом вы можете запустить средство RepAdmin.exe из PowerShell. Для этого сделайте следующее:
1. Перейдите к приглашению PowerShell и введите команду
Repadmin /showrepl * /csv | ConvertFrom-Csv | Out-GridView
2. В появившейся сетке выберите Add Criteria, затем Last Failure Status и нажмите Add.
3. Выберите подчеркнутое слово голубого цвета contains в фильтре и укажите does not equal.
4. Как показано на экране 4, введите 0 в поле, так, чтобы отфильтровывалось все со значением 0 (успех) и отображались только ошибки.
 |
| Экран 4. Задание фильтра |
Теперь, когда вы знаете, как проверять статус репликации и обнаруживать ошибки, давайте посмотрим, как выявлять и устранять четыре наиболее распространенные неисправности.
Исправление ошибки AD Replication Error -2146893022
Итак, начнем с устранения ошибки -2146893022, возникающей между DC2 и DC1. Из DC1 запустите команду Repadmin для проверки статуса репликации DC2:
Repadmin /showrepl dc2
На экране 5 показаны результаты, свидетельствующие о том, что репликация перестала выполняться, поскольку возникла проблема с DC2: целевое основное имя неверно. Тем не менее, описание ошибки может указать ложный путь, поэтому приготовьтесь копать глубже.
 |
| Экран 5. Проблема с DC2 — целевое основное имя неверно |
Во-первых, следует определить, есть ли базовое подключение LDAP между системами. Для этого запустите следующую команду из DC2:
Repadmin /bind DC1
На экране 5 видно, что вы получаете сообщение об ошибке LDAP. Далее попробуйте инициировать репликацию AD с DC2 на DC1:
Repadmin /replicate dc2 dc1 «dc=root,dc=contoso,dc=com»
И на этот раз отображается та же ошибка с главным именем, как показано на экране 5. Если открыть окно Event Viewer на DC2, вы увидите событие с Event ID 4 (см. экран 6).
 |
| Экран 6. Сообщение о событии с Event ID 4 |
Выделенный текст в событии указывает на причину ошибки. Это означает, что пароль учетной записи компьютера DC1 отличается от пароля, который хранится в AD для DC1 в Центре распределения ключей – Key Distribution Center (KDC), который в данном случае запущен на DC2. Значит, следующая наша задача – определить, соответствует ли пароль учетной записи компьютера DC1 тому, что хранится на DC2. В командной строке на DC1 введите две команды:
Repadmin /showobjmeta dc1 «cn=dc1,ou=domain controllers, dc=root,dc=contoso,dc=com» > dc1objmeta1.txt
Repadmin /showobjmeta dc2 «cn=dc1,ou=domain controllers, dc=root,dc=contoso,dc=com» > dc1objmeta2.txt
Далее откройте файлы dc1objmeta1.txt и dc1objmeta2.txt, которые были созданы, и посмотрите на различия версий для dBCSPwd, UnicodePWD, NtPwdHistory, PwdLastSet и lmPwdHistory. В нашем случае файл dc1objmeta1.txt показывает версию 19, тогда как версия в файле dc1objmeta2.txt – 11. Таким образом, сравнивая эти два файла, мы видим, что DC2 содержит информацию о старом пароле для DC1. Операция Kerberos не удалась, потому что DC1 не смог расшифровать билет службы, представленный DC2.
KDC, запущенный на DC2, не может быть использован для Kerberos вместе с DC1, так как DC2 содержит информацию о старом пароле. Чтобы решить эту проблему, вы должны заставить DC2 использовать KDC на DC1, чтобы завершить репликацию. Для этого вам, в первую очередь, необходимо остановить службу KDC на DC2:
Net stop kdc
Теперь требуется начать репликацию корневого раздела Root:
Repadmin /replicate dc2 dc1 «dc=root,dc=contoso,dc=com»
Следующим вашим шагом будет запуск двух команд Repadmin /showobjmeta снова, чтобы убедиться в том, что версии совпадают. Если все хорошо, вы можете перезапустить службу KDC:
Net start kdc
Обнаружение и устранение ошибки AD Replication Error 1908
Теперь, когда мы устранили ошибку -2146893022, давайте перейдем к ошибке репликации AD 1908, где DC1, DC2 и TRDC1 так и не удалось выполнить репликацию из ChildDC1. Решить проблему можно следующим образом. Используйте Nltest.exe для создания файла Netlogon.log, чтобы выявить причину ошибки 1908. Прежде всего, включите расширенную регистрацию на DC1, запустив команду:
Nltest /dbflag:2080fff
Теперь, когда расширенная регистрация включена, запустите репликацию между DC – так все ошибки будут зарегистрированы. Этот шаг поможет запустить три команды для воспроизведения ошибок. Итак, во-первых, запустите следующую команду на DC1:
Repadmin /replicate dc1 childdc1 dc=child,dc=root, dc=contoso,dc=com
Результат, показанный на экране 7, говорит о том, что репликация не состоялась, потому что DC домена не может быть найден.
 |
| Экран 7. Репликация не состоялась, потому что DC домена не может быть найден |
Во-вторых, из DC1 попробуйте определить местоположение KDC в домене child.root.contoso.com с помощью команды:
Nltest /dsgetdc:child /kdc
Результаты на экране 7 свидетельствуют, что такого домена нет. В-третьих, поскольку вы не можете найти KDC, попытайтесь установить связь с любым DC в дочернем домене, используя команду:
Nltest /dsgetdc:child
В очередной раз результаты говорят о том, что нет такого домена, как показано на экране 7.
Теперь, когда вы воспроизвели все ошибки, просмотрите файл Netlogon.log, созданный в папке C:Windowsdebug. Откройте его в «Блокноте» и найдите запись, которая начинается с DSGetDcName function called. Обратите внимание, что записей с таким вызовом будет несколько. Вам нужно найти запись, имеющую те же параметры, что вы указали в команде Nltest (Dom:child и Flags:KDC). Запись, которую вы ищете, будет выглядеть так:
DSGetDcName function called: client PID=2176, Dom:child Acct:(null) Flags:KDC
Вы должны просмотреть начальную запись, равно как и последующие, в этом потоке. В таблице 2 представлен пример потока 3372. Из этой таблицы следует, что поиск DNS записи KDC SRV в дочернем домене был неудачным. Ошибка 1355 указывает, что заданный домен либо не существует, либо к нему невозможно подключиться.
Поскольку вы пытаетесь подключиться к Child.root.contoso.com, следующий ваш шаг – выполнить для него команду ping из DC1. Скорее всего, вы получите сообщение о том, что хост не найден. Информация из файла Netlogon.log и ping-тест указывают на возможные проблемы в делегировании DNS. Свои подозрения вы можете проверить, сделав тест делегирования DNS. Для этого выполните следующую команду на DC1:
Dcdiag /test:dns /dnsdelegation > Dnstest.txt
На экране 8 показан пример файла Dnstest.txt. Как вы можете заметить, это проблема DNS. Считается, что IP-адрес 192.168.10.1 – адрес для DC1.
 |
| Экран 8. Пример файла Dnstest.txt |
Чтобы устранить проблему DNS, сделайте следующее:
1. На DC1 откройте консоль управления DNS.
2. Разверните Forward Lookup Zones, разверните root.contoso.com и выберите child.
3. Щелкните правой кнопкой мыши (как в родительской папке) на записи Name Server и выберите пункт Properties.
4. Выберите lamedc1.child.contoso.com и нажмите кнопку Remove.
5. Выберите Add, чтобы можно было добавить дочерний домен сервера DNS в настройки делегирования.
6. В окне Server fully qualified domain name (FQDN) введите правильный сервер childdc1.child.root.contoso.com.
7. В окне IP Addresses of this NS record введите правильный IP-адрес 192.168.10.11.
8. Дважды нажмите кнопку OK.
9. Выберите Yes в диалоговом окне, где спрашивается, хотите ли вы удалить связующую запись (glue record) lamedc1.child.contoso.com [192.168.10.1]. Glue record – это запись DNS для полномочного сервера доменных имен для делегированной зоны.
10. Используйте Nltest.exe для проверки, что вы можете найти KDC в дочернем домене. Примените опцию /force, чтобы кэш Netlogon не использовался:
Nltest /dsgetdc:child /kdc /force
11. Протестируйте репликацию AD из ChildDC1 на DC1 и DC2. Это можно сделать двумя способами. Один из них – выполнить команду
Repadmin /replicate dc1 childdc1 «dc=child,dc=root, dc=contoso,dc=com»
Другой подход заключается в использовании оснастки Active Directory Sites и Services консоли Microsoft Management Console (MMC), в этом случае правой кнопкой мыши щелкните DC и выберите Replicate Now, как показано на экране 9. Вам нужно это сделать для DC1, DC2 и TRDC1.
 |
| Экран 9. Использование оснастки Active Directory Sites и?Services |
После этого вы увидите диалоговое окно, как показано на экране 10. Не учитывайте его, нажмите OK. Я вкратце расскажу об этой ошибке.
 |
| Экран 10. Ошибка при репликации |
Когда все шаги выполнены, вернитесь к AD Replication Status Tool и обновите статус репликации на уровне леса. Ошибки 1908 больше быть не должно. Ошибка, которую вы видите, это ошибка 8606 (недостаточно атрибутов для создания объекта), как отмечалось на экране 10. Это следующая трудность, которую нужно преодолеть.
Устранение ошибки AD Replication Error 8606
Устаревший объект (lingering object) – это объект, который присутствует на DC, но был удален на одном или нескольких других DC. Ошибка репликации AD 8606 и ошибка 1988 в событиях Directory Service – хорошие индикаторы устаревших объектов. Важно учитывать, что можно успешно завершить репликацию AD и не регистрировать ошибку с DC, содержащего устаревшие объекты, поскольку репликация основана на изменениях. Если объекты не изменяются, то реплицировать их не нужно. По этой причине, выполняя очистку устаревших объектов, вы допускаете, что они есть у всех DC (а не только DCs logging errors).
Чтобы устранить проблему, в первую очередь убедитесь в наличии ошибки, выполнив следующую команду Repadmin на DC1:
Repadmin /replicate dc1 dc2 «dc=root,dc=contoso,dc=com»
Вы увидите сообщение об ошибке, как показано на экране 11. Кроме того, вы увидите событие с кодом в Event Viewer DC1 (см. экран 12). Обратите внимание, что событие с кодом 1988 только дает отчет о первом устаревшем объекте, который вам вдруг встретился. Обычно таких объектов много.
 |
| Экран 11. Ошибка из-за наличия устаревшего объекта |
 |
| Экран 12. Событие с кодом 1988 |
Вы должны скопировать три пункта из информации об ошибке 1988 в событиях: идентификатор globally unique identifier (GUID) устаревшего объекта, сервер-источник (source DC), а также уникальное, или различающееся, имя раздела – distinguished name (DN). Эта информация позволит определить, какой DC имеет данный объект.
Прежде всего, используйте GUID объекта (в данном случае 5ca6ebca-d34c-4f60-b79c-e8bd5af127d8) в следующей команде Repadmin, которая отправляет результаты в файл Objects.txt:
Repadmin /showobjmeta * «e8bd5af127d8>» > Objects.txt
Если вы откроете файл Objects.txt, то увидите, что любой DC, который возвращает метаданные репликации для данного объекта, содержит один или более устаревших объектов. DC, не имеющие копии этого объекта, сообщают статус 8439 (уникальное имя distinguished name, указанное для этой операции репликации, недействительно).
Затем вам нужно, используя GUID объект Directory System Agent (DSA) DC1, идентифицировать все устаревшие объекты в разделе Root на DC2. DSA предоставляет доступ к физическому хранилищу информации каталога, находящейся на жестком диске. В AD DSA – часть процесса Local Security Authority. Для этого выполните команду:
Repadmin /showrepl DC1 > Showrepl.txt
В Showrepl.txt GUID объект DSA DC1 появляется вверху файла и выглядит следующим образом:
DSA object GUID: 70ff33ce-2f41-4bf4-b7ca-7fa71d4ca13e
Ориентируясь на эту информацию, вы можете применить следующую команду, чтобы удостовериться в существовании устаревших объектов на DC2, сравнив его копию раздела Root с разделом Root DC1.
Repadmin /removelingeringobjects DC2 70ff33ce-2f41-4bf4- b7ca-7fa71d4ca13e «dc=root,dc=contoso,dc=com» /Advisory_mode
Далее вы можете просмотреть журнал регистрации событий Directory Service на DC2, чтобы узнать, есть ли еще какие-нибудь устаревшие объекты. Если да, то о каждом будет сообщаться в записи события 1946. Общее число устаревших объектов для проверенного раздела будет отмечено в записи события 1942.
Вы можете удалить устаревшие объекты несколькими способами. Предпочтительно использовать ReplDiag.exe. В качестве альтернативы вы можете выбрать RepAdmin.exe.
Используем ReplDiag.exe. С вашей рабочей станции администратора в корневом домене леса, а в нашем случае это Win8Client, вы должны выполнить следующие команды:
Repldiag /removelingeringobjects Repadmin /replicate dc1 dc2 «dc=root,dc=contoso,dc=com»
Первая команда удаляет объекты. Вторая команда служит для проверки успешного завершения репликации (иными словами, ошибка 8606 больше не регистрируется). Возвращая команды Repadmin /showobjmeta, вы можете убедиться в том, что объект был удален из всех, что объект был удален DC. Если у вас есть контроллер только для чтения read-only domain controller (RODC) и он содержал данный устаревший объект, вы заметите, что он все еще там находится. Дело в том, что текущая версия ReplDiag.exe не удаляет объекты из RODC. Для очистки RODC (в нашем случае, ChildDC2) выполните команду:
Repadmin /removelingeringobjects childdc2.child.root. contoso.com 70ff33ce-2f41-4bf4-b7ca-7fa71d4ca13e «dc=root,dc=contoso,dc=com» /Advisory_mode
После этого просмотрите журнал событий Directory Service на ChildDC2 и найдите событие с кодом 1939. На экране 13 вы видите уведомление о том, что устаревшие объекты были удалены.
 |
| Экран 13. Сообщение об удалении устаревших объектов |
Используем RepAdmin.exe. Другой способ, позволяющий удалить устаревшие объекты – прибегнуть к помощи RepAdmin.exe. Сначала вы должны удалить устаревшие объекты главных контроллеров домена (reference DC) с помощью кода, который видите в листинге 1. После этого необходимо удалить устаревшие объекты из всех остальных контроллеров домена (устаревшие объекты могут быть показаны или на них могут обнаружиться ссылки на нескольких контроллерах домена, поэтому убедитесь, что вы удалили их все). Необходимые для этой цели команды приведены в листинге 2.
Как видите, использовать ReplDiag.exe гораздо проще, чем RepAdmin.exe, поскольку вводить команд вам придется намного меньше. Ведь чем больше команд, тем больше шансов сделать опечатку, пропустить команду или допустить ошибку в командной строке.
Устранение ошибки AD Replication Error 8453
Предыдущие ошибки репликации AD были связаны с невозможностью найти другие контроллеры домена. Ошибка репликации AD с кодом состояния 8453 возникает, когда контроллер домена видит другие DC, но не может установить с ними связи репликации.
Например, предположим, что ChildDC2 (RODC) в дочернем домене не уведомляет о себе как о сервере глобального каталога – Global Catalog (GC). Для получения статуса ChildDC2 запустите следующие команды на ChildDC2:
Repadmin /showrepl childdc2 > Repl.txt
Данная команда отправляет результаты Repl.txt. Если вы откроете этот текстовый файл, то увидите вверху следующее:
BoulderChildDC2 DSA Options: IS_GC DISABLE_OUTBOUND_REPL IS_RODC WARNING: Not advertising as a global catalog
Если вы внимательно посмотрите на раздел Inbound Neighbors, то увидите, что раздел DC=treeroot,DC=fabrikam,DC=com отсутствует, потому что он не реплицируется. Взгляните на кнопку файла – вы увидите ошибку:
Source: BoulderTRDC1 ******* 1 CONSECTUTIVE FAILURES since 2014-01-12 11:24:30 Last error: 8453 (0x2105): Replication access was denied Naming Context: DC=treeroot,DC=fabrikam,DC=com
Эта ошибка означает, что ChildDC2 не может добавить связь репликации (replication link) для раздела Treeroot. Как показано на экране 14, данная ошибка также записывается в журнал регистрации событий Directory Services на ChildDC2 как событие с кодом 1926.
 |
| Экран 14. Отсутствие связи репликации |
Здесь вам нужно проверить, нет ли проблем, связанных с безопасностью. Для этого используйте DCDiag.exe:
Dcdiag /test:checksecurityerror
На экране 15 показан фрагмент вывода DCDiag.exe.
 |
| Экран 15. Фрагмент вывода DCDiag.exe |
Как видите, вы получаете ошибку 8453, потому что группа безопасности Enterprise Read-Only Domain Controllers не имеет разрешения Replicating Directory Changes.
Чтобы решить проблему, вам нужно добавить отсутствующую запись контроля доступа – missing access control entry (ACE) в раздел Treeroot. В этом вам помогут следующие шаги:
1. На TRDC1 откройте оснастку ADSI Edit.
2. Правой кнопкой мыши щелкните DC=treeroot,DC=fabrikam,DC=com и выберите Properties.
3. Выберите вкладку Security.
4. Посмотрите разрешения на этот раздел. Отметьте, что нет записей для группы безопасности Enterprise Read-Only Domain Controllers.
5. Нажмите Add.
6. В окне Enter the object names to select наберите ROOTEnterprise Read-Only Domain Controllers.
7. Нажмите кнопку Check Names, затем выберите OK, если указатель объектов (object picker) разрешает имя.
8. В диалоговом окне Permissions для Enterprise Read-Only Domain Controllers снимите флажки Allow для следующих разрешений
*Read
*Read domain password & lockout policies («Чтение политики блокировки и пароля домена»)
*Read Other domain parameters
9. Выберите флажок Allow для разрешения Replicating Directory Changes, как показано на экране 16. Нажмите OK.
10. Вручную запустите Knowledge Consistency Checker (KCC), чтобы немедленно сделать перерасчет топологии входящей репликации на ChildDC2, выполнив команду
Repadmin /kcc childdc2
 |
| Экран 16. Включение разрешения Replicating Directory Change |
Данная команда заставляет KCC на каждом целевом сервере DC незамедлительно делать перерасчет топологии входящей репликации, добавляя снова раздел Treeroot.
Состояние репликации критически важно
Репликация во всех отношениях в лесу AD имеет решающее значение. Следует регулярно проводить ее диагностику, чтобы изменения были видны всем контроллерам домена, иначе могут возникать различные проблемы, в том числе связанные с аутентификацией. Проблемы репликации нельзя обнаружить сразу. Поэтому если вы пренебрегаете мониторингом репликации (в крайнем случае, периодически делайте проверку), то рискуете столкнуться с трудностями в самый неподходящий момент. Моей задачей было показать вам, как проверять статус репликации, обнаруживать ошибки и в то же время как справиться с четырьмя типичными проблемами репликации AD.
Листинг 1. Команды для удаления устаревших объектов из Reference DC
REM Команды для удаления устаревших объектов REM из раздела Configuration. Repadmin /removelingeringobjects childdc1.child.root. contoso.com 70ff33ce-2f41-4bf4-b7ca-7fa71d4ca13e «cn=configuration,dc=root,dc=contoso,dc=com» Repadmin /removelingeringobjects childdc1.child.root. contoso.com 3fe45b7f-e6b1-42b1-bcf4-2561c38cc3a6 «cn=configuration,dc=root,dc=contoso,dc=com» Repadmin /removelingeringobjects childdc1.child.root. contoso.com 0b457f73-96a4-429b-ba81-1a3e0f51c848 «cn=configuration,dc=root,dc=contoso,dc=com» REM Команды для удаления устаревших объектов REM из раздела ForestDNSZones. Repadmin /removelingeringobjects childdc1.child.root. contoso.com 70ff33ce-2f41-4bf4-b7ca-7fa71d4ca13e «dc=forestdnszones,dc=root,dc=contoso,dc=com» Repadmin /removelingeringobjects childdc1.child.root. contoso.com 3fe45b7f-e6b1-42b1-bcf4-2561c38cc3a6 «dc=forestdnszones,dc=root,dc=contoso,dc=com» Repadmin /removelingeringobjects childdc1.child. root.contoso.com 0b457f73-96a4-429b-ba81- 1a3e0f51c848 «dc=forestdnszones,dc=root, dc=contoso,dc=com» REM Команды для удаления устаревших объектов REM из раздела домена Root. Repadmin /removelingeringobjects dc1.root. contoso.com 3fe45b7f-e6b1-42b1-bcf4-2561c38cc3a6 «dc=root,dc=contoso,dc=com» REM Команды для удаления устаревших объектов REM из раздела DomainDNSZones. Repadmin /removelingeringobjects dc1.root. contoso.com 3fe45b7f-e6b1-42b1-bcf4-2561c38cc3a6 «dc=root,dc=contoso,dc=com»
Листинг 2. Команды для удаления устаревших объектов из остальных DC
REM Команды для удаления устаревших объектов REM из раздела Configuration. Repadmin /removelingeringobjects dc1.root. contoso.com 0c559ee4-0adc-42a7-8668-e34480f9e604 «cn=configuration,dc=root,dc=contoso,dc=com» Repadmin /removelingeringobjects dc2.root. contoso.com 0c559ee4-0adc-42a7-8668-e34480f9e604 «cn=configuration,dc=root,dc=contoso,dc=com» Repadmin /removelingeringobjects childdc2.child.root. contoso.com 0b457f73-96a4-429b-ba81-1a3e0f51c848 «cn=configuration,dc=root,dc=contoso,dc=com» Repadmin /removelingeringobjects trdc1.treeroot. fabrikam.com 0c559ee4-0adc-42a7-8668-e34480f9e604 «cn=configuration,dc=root,dc=contoso,dc=com» REM Команды для удаления устаревших объектов REM из раздела ForestDNSZones. Repadmin /removelingeringobjects dc1.root.contoso. com 0c559ee4-0adc-42a7-8668-e34480f9e604 «dc=forestdnszones,dc=root,dc=contoso,dc=com» Repadmin /removelingeringobjects dc2.root.contoso. com 0c559ee4-0adc-42a7-8668-e34480f9e604 «dc=forestdnszones,dc=root,dc=contoso,dc=com» Repadmin /removelingeringobjects childdc2.child.root. contoso.com 0b457f73-96a4-429b-ba81-1a3e0f51c848 «dc=forestdnszones,dc=root,dc=contoso,dc=com» Repadmin /removelingeringobjects trdc1.treeroot. fabrikam.com 0c559ee4-0adc-42a7-8668-e34480f9e604 «dc=forestdnszones,dc=root,dc=contoso,dc=com» REM Команды для удаления устаревших объектов REM из раздела DomainDNSZones–Root. Repadmin /removelingeringobjects dc2.child.root. contoso.com 70ff33ce-2f41-4bf4-b7ca-7fa71d4ca13e «dc=domaindnszones,dc=root,dc=contoso,dc=com» REM Команды для удаления устаревших объектов REM из раздела домена Child. Repadmin /removelingeringobjects dc1.root.contoso. com 0c559ee4-0adc-42a7-8668-e34480f9e604 «dc=child,dc=root,dc=contoso,dc=com» Repadmin /removelingeringobjects dc2.root.contoso. com 0c559ee4-0adc-42a7-8668-e34480f9e604 «dc=child,dc=root,dc=contoso,dc=com» Repadmin /removelingeringobjects childdc2.child.root. contoso.com 0b457f73-96a4-429b-ba81-1a3e0f51c848 «dc=child,dc=root,dc=contoso,dc=com» Repadmin /removelingeringobjects trdc1.treeroot. fabrikam.com 0c559ee4-0adc-42a7-8668-e34480f9e604 «dc=child,dc=root,dc=contoso,dc=com» REM Команды для удаления устаревших объектов REM из раздела DomainDNSZones-Child. Repadmin /removelingeringobjects childdc2.child.root. contoso.com 0c559ee4-0adc-42a7-8668-e34480f9e604 «dc=domaindnszones,dc=child,dc=root,dc=contoso,dc=com» REM Команды для удаления устаревших объектов REM из раздела домена TreeRoot. Repadmin /removelingeringobjects childdc1.child.root. contoso.com 0b457f73-96a4-429b-ba81-1a3e0f51c848 «dc=treeroot,dc=fabrikam,dc=com» Repadmin /removelingeringobjects childdc2.child.root. contoso.com 0b457f73-96a4-429b-ba81-1a3e0f51c848 «dc=treeroot,dc=fabrikam,dc=com» Repadmin /removelingeringobjects dc1.root.contoso.com 0b457f73-96a4-429b-ba81-1a3e0f51c848 «dc=treeroot,dc=fabrikam,dc=com» Repadmin /removelingeringobjects dc2.root.contoso.com 0b457f73-96a4-429b-ba81-1a3e0f51c848 «dc=treeroot,dc=fabrikam,dc=com»

сайтов и блокирует действие белка DnaB (хеликаза) — препятствует раскручиванию двойной спирали ДНК.
4.5 Репликация у эукариот
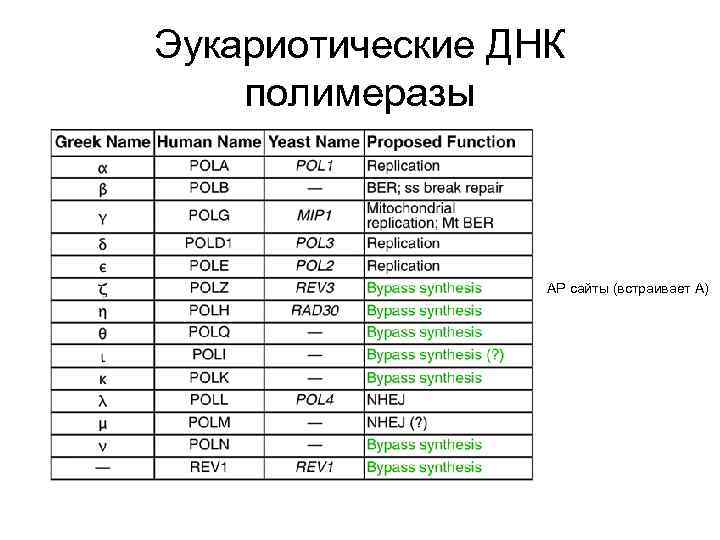
Эукариоты содержат более 10 ДНК-полимераз, которые объединяют в 6 семейств: A, B, C, D, X и Y. Сами полимеразы обычно называют греческими буквами. ДНК-полимераза α синтезирует праймеры, высокопроцессивная ДНК-полимераза δ синтезирует запаздывающую цепь, ДНК-полимераза ε синтезирует лидирующую цепь и может заменить ДНК-полимеразу δ в синтезе запаздывающей цепи. ДНКполимераза β участвует в репарации ДНК. Скорость репликации у эукариот в 10 раз ниже, чем у прокариот. Эукариотическая ДНК имеет множество точек ori. У эукариот имеются аналоги бактериальных белков, участвующих в репликации:
1.Белковый MCM-комплекс выполняет роль хеликазы — аналогичен
DnaA.
2.RPA-белки (replication protein A) связываются с цепями ДНК подобно SSB-белкам у прокариот.
3.Ферменты РНКаза H1 и FEN1 (flap endonuclease-1) действуют как прокариотическая ДНК-полимераза I (т.е. вырезают праймер).
4.Белок PCNA (пролиферирующий клеточный ядерный антиген) является аналогом «β -хомута» прокариот.
АПроблема образования супервитков ДНК
Входе репликации возникают положительные супервитки и торсионное напряжение на цепях ДНК, которые снимаются ферментами ДНК-гиразами (ДНКтопоизомеразами II). Это необходимо для того, чтобы полимераза могла продолжать движение по цепи ДНК — супервитки этому препятствуют.
Б Проблема высокой точности процесса
Точность самой полимеразной реакции была определена в ходе исследований in vitro. ДНК-полимераза совершает 1 ошибку на каждые 104–105 нуклеотидов. Т.е. 1 нуклеотид из 10 000 оказывается некомплементарным нуклеотиду в родительской цепи.
Исследованиями было доказано, что общая частота ошибок репликации у кишечной палочки E. coli составляет 1 на каждые 109–1010 нуклеотидов. Такой хороший показатель достигается двумя основными факторами (в комплексе с достаточно низкой частотой ошибок самой ДНК-полимеразы):
1.После каждого цикла полимеразной реакции ДНК-полимераза «определяет», образовалась ли комплементарная пара между новым нуклеотидом в дочерней цепи и нуклеотидом родительской. Если нет, то она «делает шаг назад» и вырезает только что присоединённый некомплементарный нуклеотид. Иными словами, ДНК-полимераза проявляет

Проблемы репликации 91
3’→5’ экзонуклеазную активность. Она позволяет снизить обычную частоту ошибок полимеразы с 1 на каждые 104–105 нуклеотидов до 1 на каждые 106–108.
2.Однако и этого недостаточно, чтобы объяснить ещё более высокую точность репликации, которая отмечается in vivo. Дополнительным фактором служит отдельная система репарации — комплекс ферментов, которые «сканируют» цепи ДНК на наличие ошибок и исправляют их в случае обнаружения.
ВПроблема репликации генома у эукариот
Как правило, геном одной эукариотической клетки состоит из множества молекул ДНК общей длиной до нескольких миллиардов нуклеотидных пар (в каждой соматической ядерной клетке человека — 23 пары хромосом — 3,2 миллиарда нуклеотидов).
Скорость репликации у эукариот невысока (~100 нуклеотидов в секунду), поэтому репликация такого объёма ДНК заняла бы месяцы или даже годы. Эта проблема решается множественностью точек ori. Т.е. репликация у эукариот начинается сразу во многих точках и идёт параллельно.
ГПроблема репликации 3’-конца родительской цепи ДНК
ДНК-полимераза не может полностью реплицировать 3’-конец родительской цепи. Это объясняется тем, что праймер, присоединённый к 3’-концу, разрушается ферментами, удаляющими любые рибонуклеотиды, связанные с ДНК. Для этого эукариоты выработали особые структуры, позволяющие защитить ДНК от укорочения — теломеры. Они представляют собой последовательности нуклеотидных остатков на концах ДНК, состоящие из 300 и более нуклеотидов (у людей — несколько килобаз повторов TTAGGG). Теломеры синтезируются особым ферментом теломеразой (комплекс белка и РНК). Фрагмент РНК, входящий в состав теломеразы, комплементарен теломерной последовательности нуклеотидов и служит матрицей в их синтезе. Теломеразы активны только в стволовых клетках и некоторых лейкоцитах. В других клетках эти ферменты не активны, и удлинения теломер не происходит.
В каждом цикле репликации теломеры на концах ДНК укорачиваются примерно на 50–250 нуклеотидов. Как только укорочение теломер становится критическим, клетка может пойти по одному из нескольких сценариев: например, может начаться процесс программируемой клеточной гибели — апоптоз.
Как правило, теломеры отсутствуют у большинства прокариот — их ДНК кольцевая, поэтому необходимости в них нет. Однако у бактерий родов Streptomyces и Borrelia, имеющих линейную ДНК, они есть. При этом они отличаются по структуре и функциям от эукариотических.
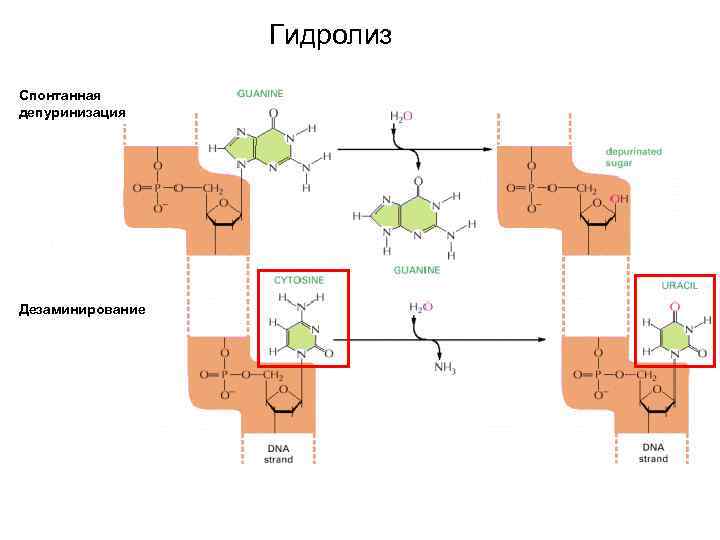
Причины появления повреждений в ДНК • Ошибки репликации • Повреждения ДНК эндогенными агентами Гидролиз (депуринизация, дезаминирование) • Повреждения ДНК экзогенными агентами облучение повреждение химическими агентами (например, алкилирование) • Репликация «через повреждения» с использованием полимераз, отличающихся низкой точностью копирования
Гидролиз Спонтанная депуринизация Дезаминирование
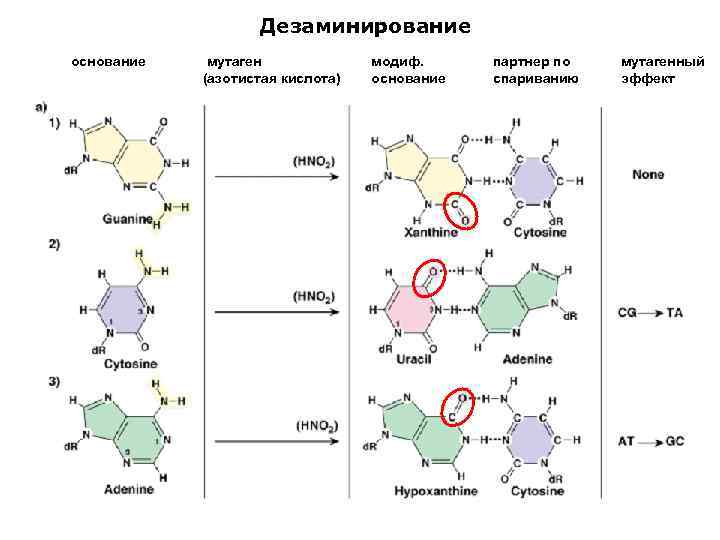
Дезаминирование основание мутаген (азотистая кислота) модиф. основание партнер по спариванию мутагенный эффект
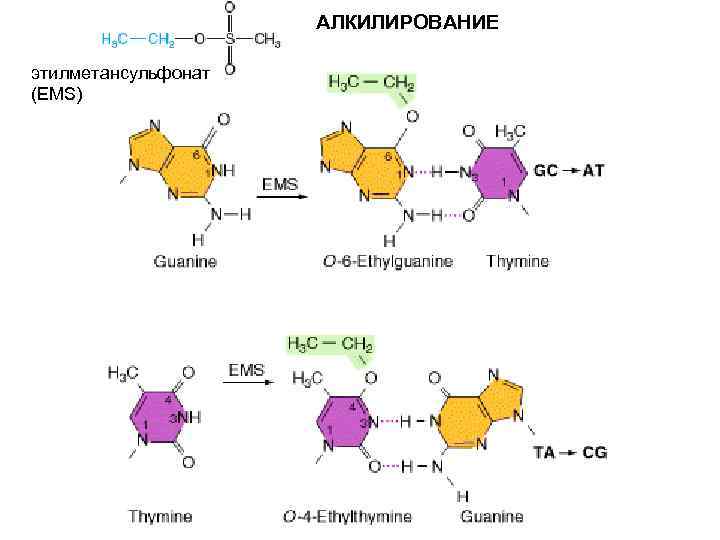
АЛКИЛИРОВАНИЕ этилметансульфонат (EMS)
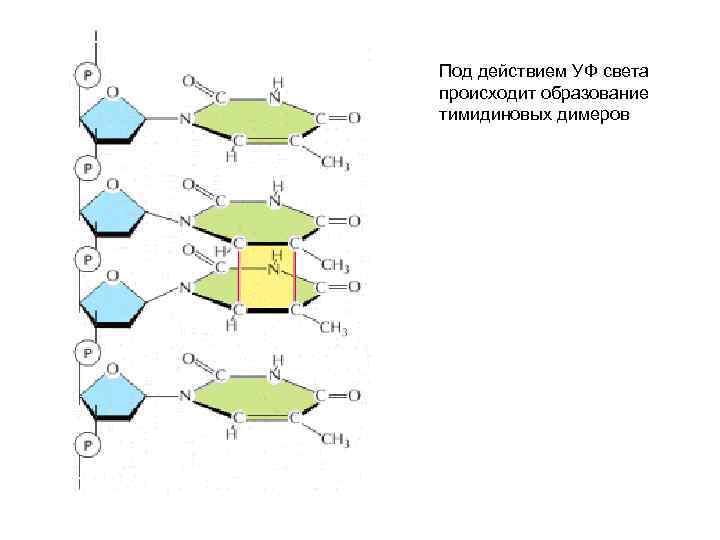
Под действием УФ света происходит образование тимидиновых димеров
Cтратегии коррекции повреждений • Ошибки репликации: • • исправление ошибок ДНК полимеразой (3’-5’ экзонуклеаза), репарация неспаренных оснований (mismatch repair) • Повреждение ДНК эндогенными и экзогенными агентами: • • Прямое удаление повреждений Эксцизия оснований (base excision repair) Эксцизия нуклеотидов (nucleotide excision repair) Рекомбинация и черезблоковый синтез особыми полимеразами (не удаляет ошибок но позволяет продолжить репликацию)
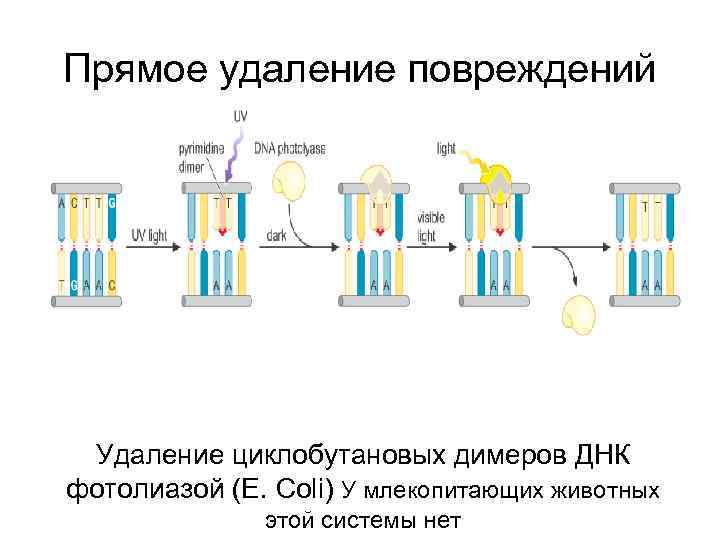
Прямое удаление повреждений Удаление циклобутановых димеров ДНК фотолиазой (E. Coli) У млекопитающих животных этой системы нет
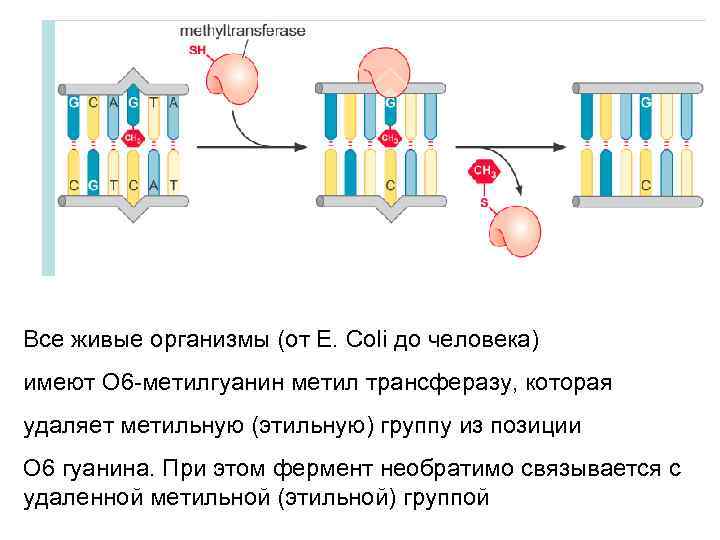
Все живые организмы (от E. Coli до человека) имеют О 6 -метилгуанин метил трансферазу, которая удаляет метильную (этильную) группу из позиции О 6 гуанина. При этом фермент необратимо связывается с удаленной метильной (этильной) группой
Другие повреждения удаляются с помощью более сложных механизмов Эксцизия оснований (base excision repair) удаляется один поврежденный нуклеотид Эксцизия нуклеотидов (nucleotide excision repair) удаляется достаточно протяженный олигонуклеотид (12 -13 н. у E. Coli и 24 -32 н. у эукариотических организмов)
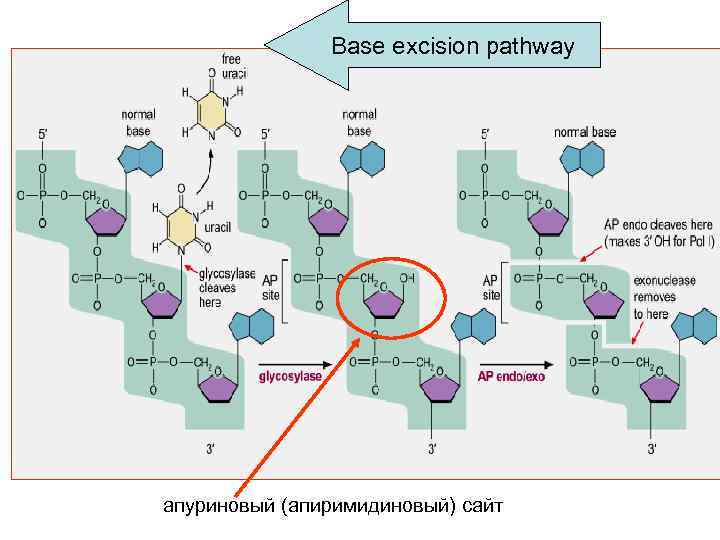
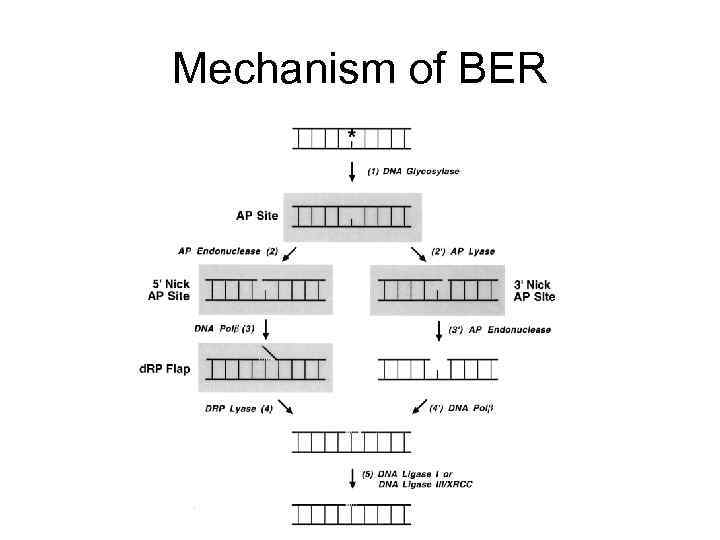
Base excision repair (BER) эксцизия (вырезание) оснований прокариоты и эукариоты • Основной путь удаления модифицированных (в том числе окисленных) оснований и включенного по ошибке урацила • Различные ДНК-гликозилазы адресно узнают поврежденные основания и удаляют их, разрезая гликозидную связь. При этом возникает AP (апуриновый/апиримидиновый) сайт. • В клетке существует несколько АР эндонуклеаз, которые разрезают фосфодиэфирный остов ДНК рядом с AP сайтом • AP nucleotide удаляется экзонуклеазой/дезоксирибофосфодиэстеразой и «брешь» застраивается ДНК полимеразой
Base excision pathway апуриновый (апиримидиновый) сайт
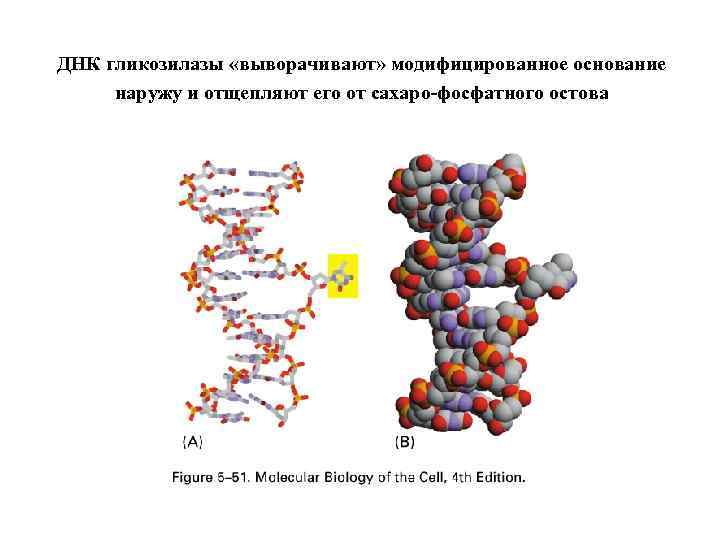
ДНК гликозилазы «выворачивают» модифицированное основание наружу и отщепляют его от сахаро-фосфатного остова
Mechanism of BER
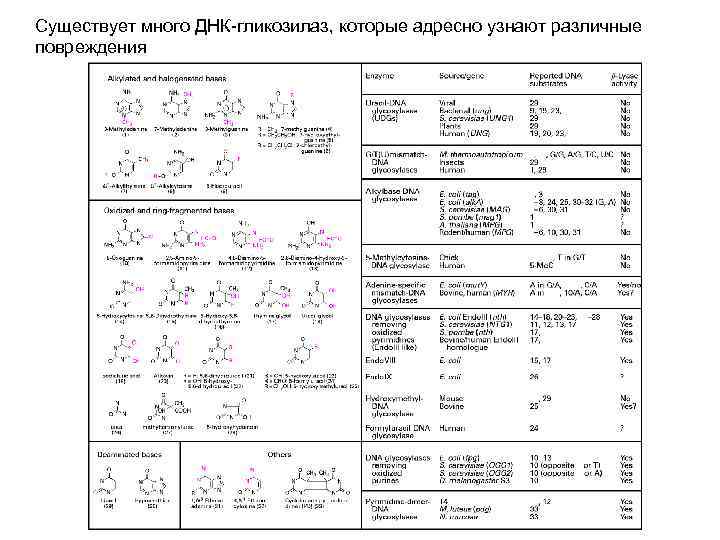
Существует много ДНК-гликозилаз, которые адресно узнают различные повреждения
ДНК-гликозизазы, присутствующие в клетках человека DNA glycosylase Specific for MBD 4 Uracil MPG Ethenoadenine, hypoxanthine, 3 methyladenine NTH 1 Cytosine glycol, dihydrouracil, formamidopyrimidine, thymine glycol OGG 1 Formamidopyrimidine, 8 -oxoguanine SMUG 1 Uracil TDG Ethenocytosine, uracil UNG Uracil, 5 -hydroxyuracil
Основные типы повреждений, которые удаляются посредством BER (большая часть не блокирует репликацию) • Окисленные основания, в том числе 8 -oкси-G, который спаривается с А, вызывая GC —> TA трансверсии • Дезоксиурацил • Различные продукты алкилирования оснований (например, 3 -me. A) • Спонтанно возникающие апуриновые сайты
Эксцизия нуклеотидов (nucleotide excision repair) прокариоты и эукриоты Этот механизм используется для коррекции «серьезных» повреждений, которые блокируют репликацию (у человека таковыми являются, в частности, тимидиновые димеры). 1. Особые белки узнают поврежденные участки ДНК и привлекают специальные нуклеазы, которые вносят разрывы на некотором расстоянии перед повреждением и на некотором расстоянии после него. 2. Фрагмент ДНК, содержащий повреждение, удаляется, и образовавшаяся брешь застраивается ДНК полимеразой
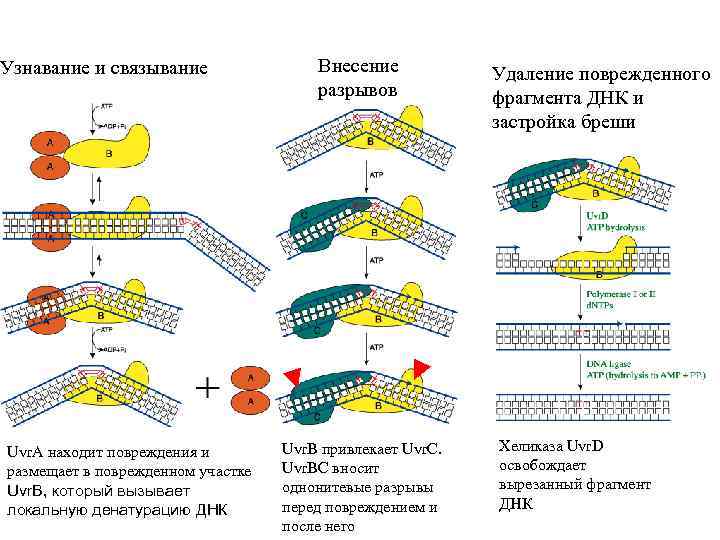
У E. Coli четыре белка участвуют в эксцизии нуклеотидов: Uvr. A, Uvr. B, Uvr. C, Uvr. D 1. Uvr. A и Uvr. B сканируют ДНК и выявляют поврежденные места 2. После выявления поврежденного участка Uvr. A освобождается из комплекса, а Uvr. B вызывает локальную денатурацию поврежденного участка и привлекает Uvr. C. 3. Комплекс Uvr. BС вносит однонитевые разрывы с 5’и 3’- конца от повреждения 4. DNA хеликаза Uvr. D обеспечивает удаление из дуплекса фрагмента ДНК, содержащего повреждение. 5. DNA Pol I застраивает брешь и лигаза «зашивает» однонитевой разрыв.
Узнавание и связывание Uvr. A находит повреждения и размещает в поврежденном участке Uvr. B, который вызывает локальную денатурацию ДНК Внесение разрывов Uvr. B привлекает Uvr. C. Uvr. BC вносит однонитевые разрывы перед повреждением и после него Удаление поврежденного фрагмента ДНК и застройка бреши Хеликаза Uvr. D освобождает вырезанный фрагмент ДНК
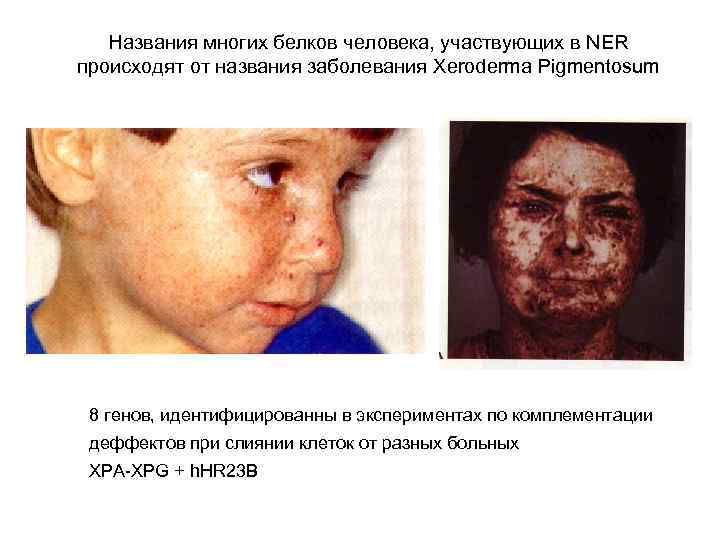
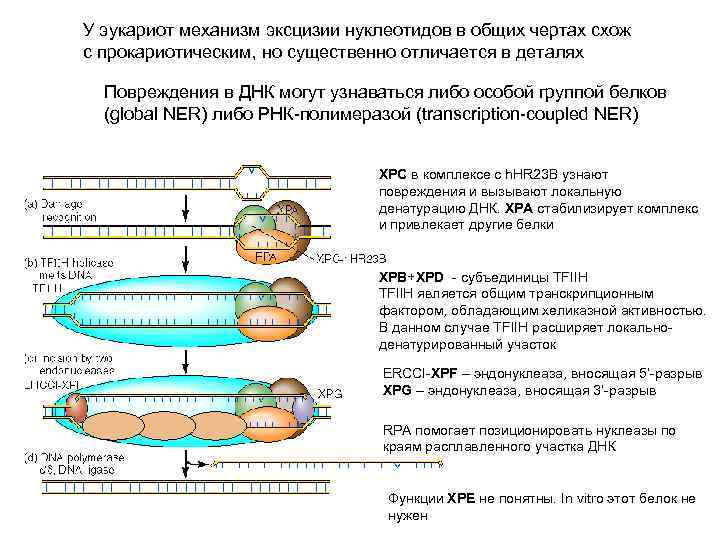
Названия многих белков человека, участвующих в NER происходят от названия заболевания Xeroderma Pigmentosum 8 генов, идентифицированны в экспериментах по комплементации деффектов при слиянии клеток от разных больных XPA-XPG + h. HR 23 B
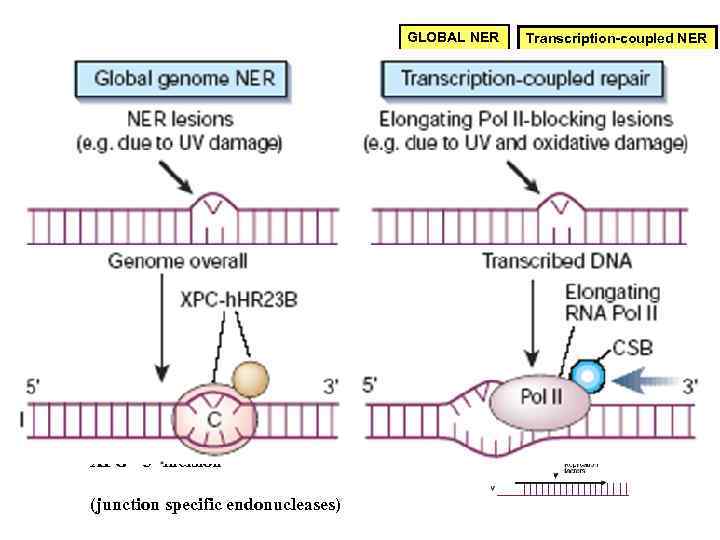
У эукариот механизм эксцизии нуклеотидов в общих чертах схож с прокариотическим, но существенно отличается в деталях Повреждения в ДНК могут узнаваться либо особой группой белков (global NER) либо РНК-полимеразой (transcription-coupled NER) XPC в комплексе с h. HR 23 B узнают повреждения и вызывают локальную денатурацию ДНК. XPA стабилизирует комплекс и привлекает другие белки XPB+XPD — субъединицы TFIIH является общим транскрипционным фактором, обладающим хеликазной активностью. В данном случае TFIIH расширяет локальноденатурированный участок ERCCI-XPF – эндонуклеаза, вносящая 5’-разрыв XPG – эндонуклеаза, вносящая 3’-разрыв RPA помогает позиционировать нуклеазы по краям расплавленного участка ДНК Функции XPE не понятны. In vitro этот белок не нужен
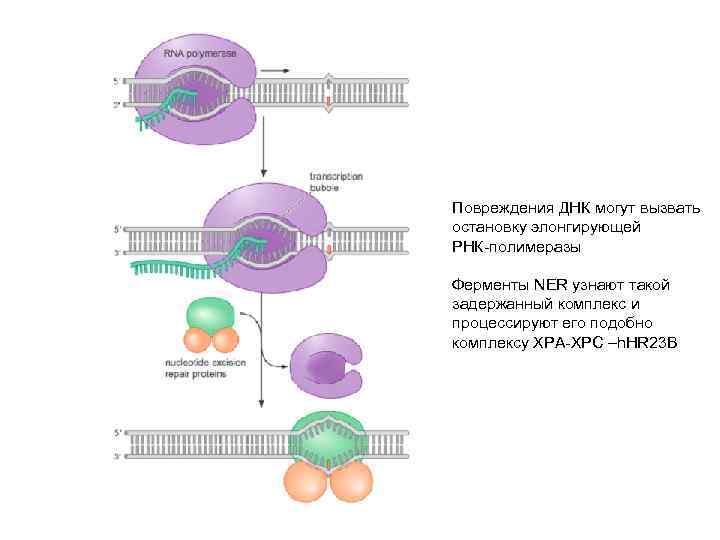
Повреждения ДНК могут вызвать остановку элонгирующей РНК-полимеразы Ферменты NER узнают такой задержанный комплекс и процессируют его подобно комплексу XPA-XPC –h. HR 23 B
GLOBAL NER Transcription-coupled NER HPC-h. HR 23 B Pol II XPC — damage recognition TFIIH XPB & XPD – TFII H DNA helicase XPG XPA stabilisation of SS DNA fragment ERCC 1 -XPF — 5’ incision XPG — 3’ incision (junction specific endonucleases) XPG
Исправление ошибок репликации (Мismatch repair) • ДНК полимеразы (даже те, у которых есть корректирующая активность) все равно делают ошибки, которые надо исправлять • Система репарации ошибок репликации должна • 1. Быстро находить ошибки • 2. Различать родительскую и новосинтезированную • цепь с тем, чтобы в неспаренном участке • заменить ошибочно включенный нуклеотид
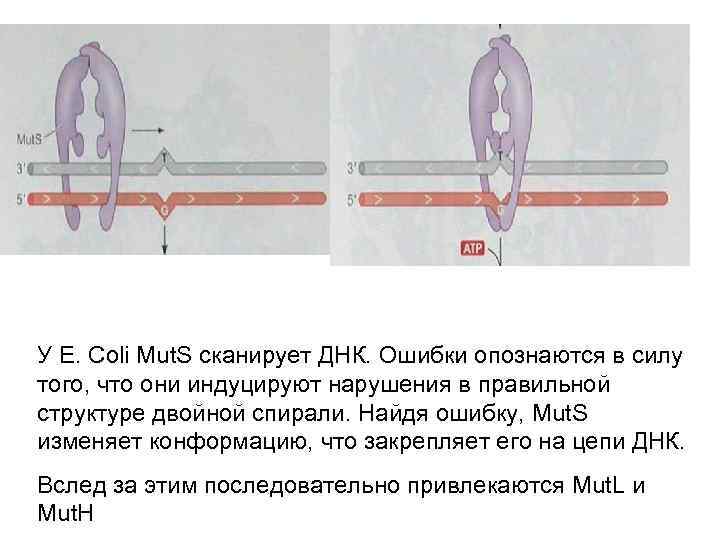
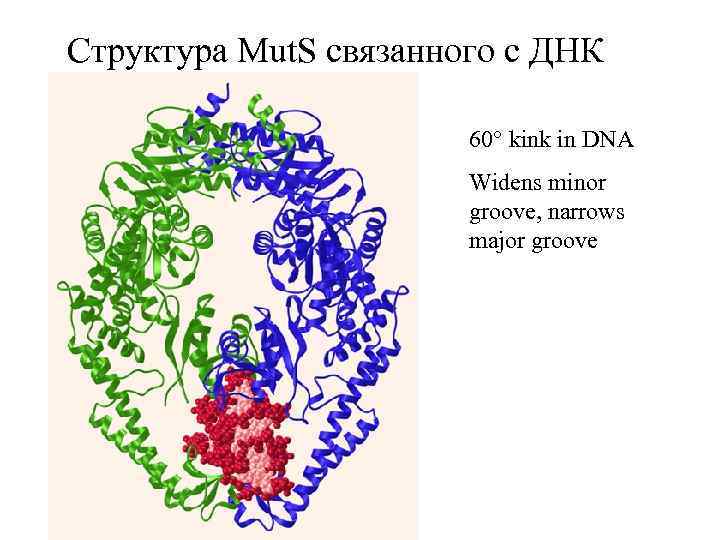
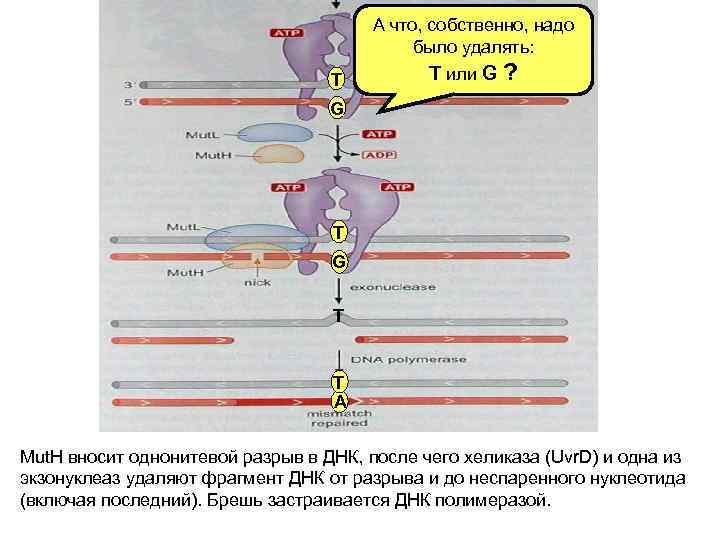
У E. Coli Mut. S сканирует ДНК. Ошибки опознаются в силу того, что они индуцируют нарушения в правильной структуре двойной спирали. Найдя ошибку, Mut. S изменяет конформацию, что закрепляет его на цепи ДНК. Вслед за этим последовательно привлекаются Mut. L и Mut. H
Структура Mut. S связанного с ДНК 60° kink in DNA Widens minor groove, narrows major groove
Что узнается ? • Mut. S димер (у дрожжей, Msh 2/Msh 3 или Msh 2/Msh 6 гетеродимер) • Эксперименты по связыванию с ДНК in vitro и репарации гетеродуплексов in vivo показали, что MMR узнает все комбинации неспаренных оснований, кроме C: C, а также короткие <4 п. н. делеции и инсерции ( «инделы» ) • неправильные пары G: T and A: C и инсерции/делеции в 1 п. особенно хорошо узнаются. Эти нарушения являются наиболее частыми ошибками ДНК-полимераз
А что, собственно, надо было удалять: T T или G ? G T T A Mut. H вносит однонитевой разрыв в ДНК, после чего хеликаза (Uvr. D) и одна из экзонуклеаз удаляют фрагмент ДНК от разрыва и до неспаренного нуклеотида (включая последний). Брешь застраивается ДНК полимеразой.
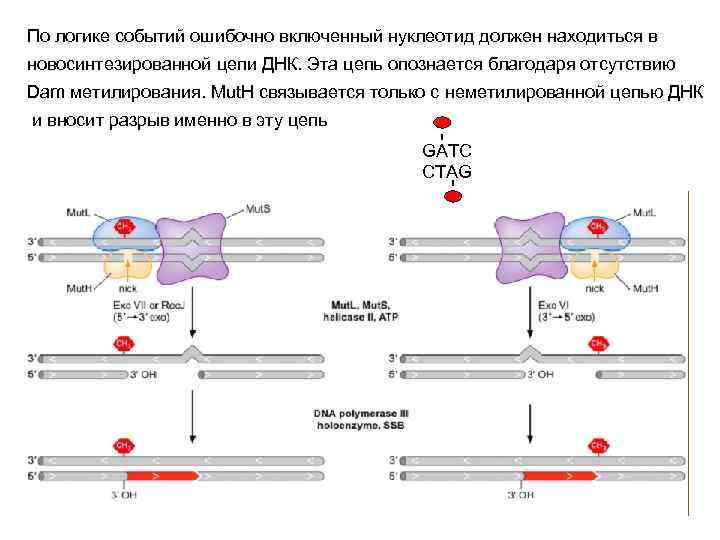
По логике событий ошибочно включенный нуклеотид должен находиться в новосинтезированной цепи ДНК. Эта цепь опознается благодаря отсутствию Dam метилирования. Mut. H связывается только с неметилированной цепью ДНК и вносит разрыв именно в эту цепь GATC CTAG
В эукариотических клетках также существует система коррекции ошибок репликации Обнаружены гомологи Mut. S и Mut. L; гомолога Mut. H не обнаружено гомологи Mut. S (MSH — Mut. S homolog) образуют два гетеродимерных комплекса — MSH 2 -MSH 6 (Mut. Sα) узнает неспаренные нуклеотиды и короткие «инделы» — MSH 2 -MSH 3 (Mut. Sβ) узнает длинные «инделы» Механизм распознавания новосинтезированной цепи не известен наличие разрывов?
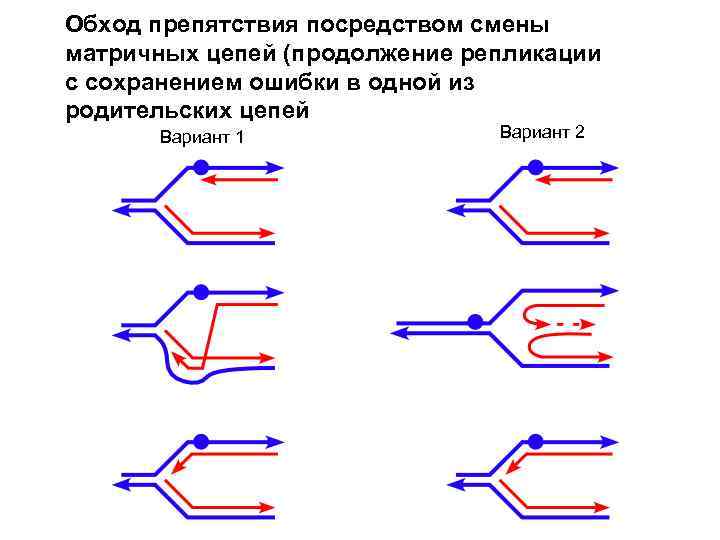
А если не успели все починить а ДНК уже реплицируется?
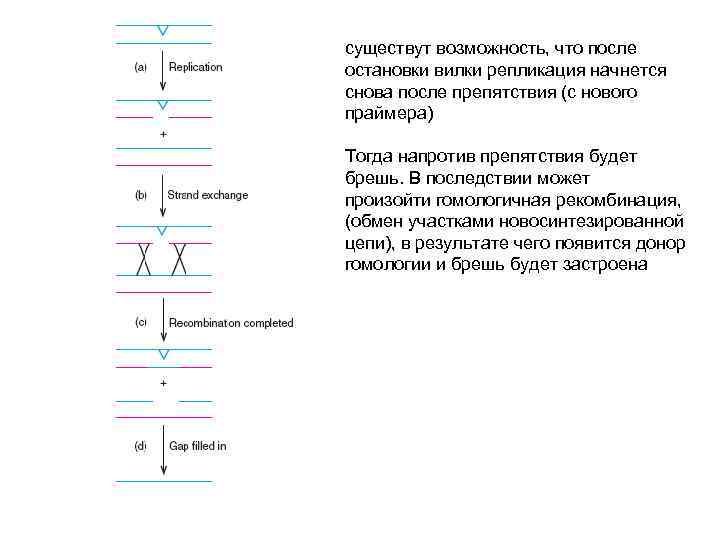
Обход препятствия посредством смены матричных цепей (продолжение репликации с сохранением ошибки в одной из родительских цепей Вариант 1 Вариант 2
существут возможность, что после остановки вилки репликация начнется снова после препятствия (с нового праймера) Тогда напротив препятствия будет брешь. В последствии может произойти гомологичная рекомбинация, (обмен участками новосинтезированной цепи), в результате чего появится донор гомологии и брешь будет застроена
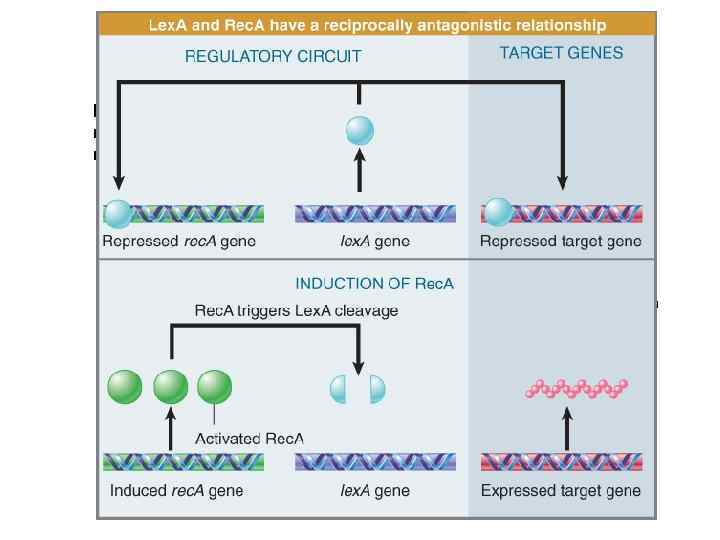
SOS репарация у Е. Сoli Прежде всего об остановке репликации надо сообщить Rec A Cвязывается с однонитевой ДНК и образует ДНК-белковые филаменты Однонитевые участки ДНК образуются при остановке репликативных вилок Lex A Мастер-регулятор транскрипции генов, кодирующих участвующие в репарации повреждений ДНК белки (31 ген или более) Димеры Lex A связываются с SOS боксами (20 п. н. консенсусы) в операторах генов репарации и ингибируют транскрипцию
Филаменты Rec A стимулируют аутопротеолиз Lex A При снижении концентрации Lex A сначала активируются, гены, контролируемые операторами, в состав которых входят низкоафинные Lex A боксы lex. A, rec. A, uvr. B, and uvr. D При дальнейшем снижении уровня Lex A активируются гены, обеспецивающие осуществление репарации с ошибками (мутазы) Umu. DC оперон
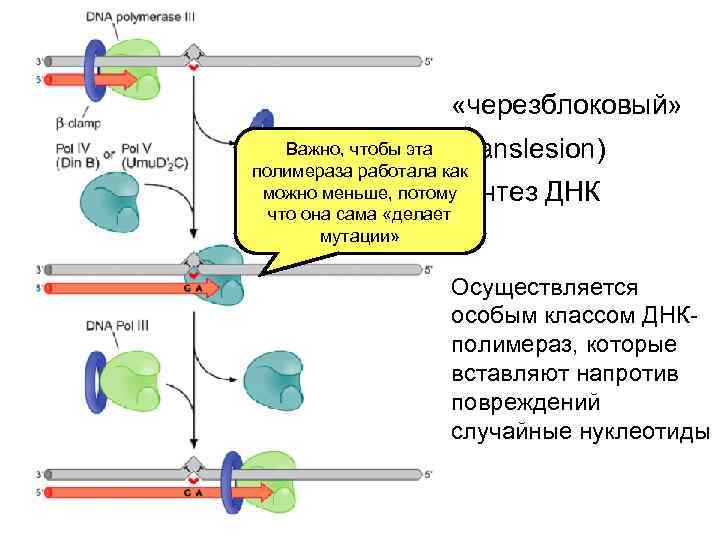
Umu. D повергается автопротеолитическому расщеплению с образованием активного фрагмента Umu. D’ активирует черезблоковую полимеразу Umu. C комплекс (Umu. D’)2 -Umu. C теперь называют ДНК полимераза V эта полимераза осуществляет репликацию через AP сайты, тимидиновые димеры и ряд других повреждений
«черезблоковый» Важно, чтобы эта (translesion) полимераза работала как можно меньше, потому синтез ДНК что она сама «делает мутации» Осуществляется особым классом ДНКполимераз, которые вставляют напротив повреждений случайные нуклеотиды
«черезблоковый» синтез катализируемой «мутасомой» (DNA pol V)” Cowcatcher model G При наличии препятствия на пути ДНК поллимеразы III, эта полимераза диссоциирует от ДНК. Хеликаза продолжает работать, генерируя однотитевой участок, с которым связывается Rec. A Pol V связывается со свободным праймером. Для эффективного связывания необходим контакт с Rec A и с бета-зажимом Непосредственно в месте повреждения с ДНК связывается SSB Locomotive Pol V начинает синтез ДНК, одновременно вытесняя Rec A cowcatcher филамент. Одновременно происходит процессивная диссоциация Rec A филамента с противоположного конца, сопряденная с (скотосбраыватель) гидролизом ATФ. Pham P et al. PNAS 2001; 98: 8350 -8354 © 2001 by The National Academy of Sciences После удаления всего филамента Rec A Pol V диссоциирует от ДНК, освобождая место для Pol III. Таким образом время работы Pol V определяется временем существования Rec A филамента. Pol V успевает включить несколько нуклеотидов. Напротив тимидинового димера ТТ чаще всего включается GA
Эукариотические ДНК полимеразы АР сайты (встраивает А)
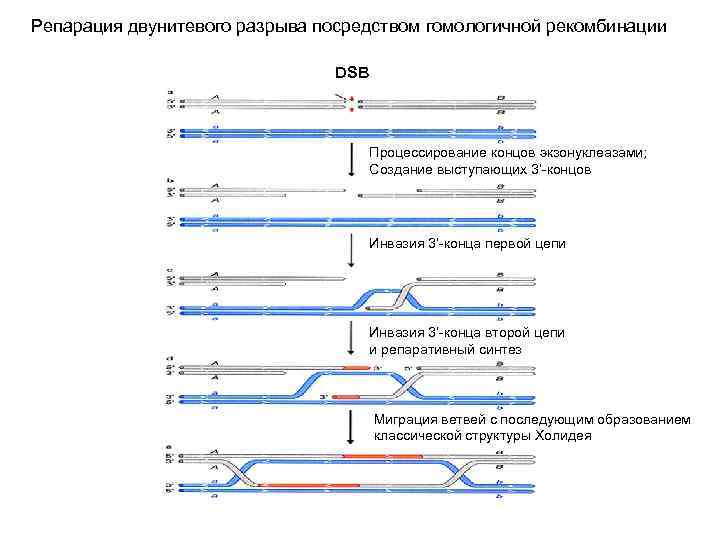
Репарация двунитевых разрывов Двунитевые разрывы в ДНК возникают: под действием ионизирующего излучения под действием некоторых химических агентов, в частности, ингибиторов ДНК топоизомеразы II Существует два основных пути репарации двунитевых разрывов: гомологичная рекомбинация негомологичное соединение концов ДНК
Репарация двунитевого разрыва посредством гомологичной рекомбинации DSB Процессирование концов экзонуклеазами; Создание выступающих 3’-концов Инвазия 3’-конца первой цепи Инвазия 3’-конца второй цепи и репаративный синтез Миграция ветвей с последующим образованием классической структуры Холидея
Для репарации двунитевых разрывов с использованием системы гомологичной рекомбинации необходимы: Донор гомологии (например гомологичная хромосома или сестринская хроматида) Белок, облегчающий инвазию цепи, и другие компоненты системы гомологичной рекомбинации
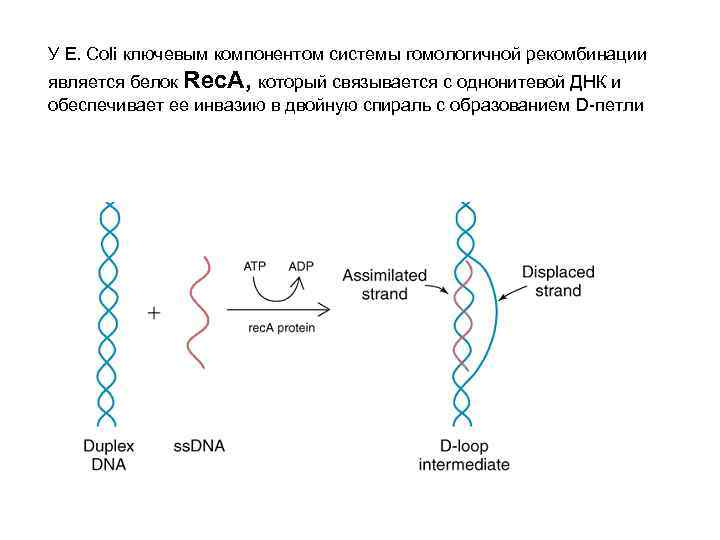
У E. Coli ключевым компонентом системы гомологичной рекомбинации является белок Rec. A, который связывается с однонитевой ДНК и обеспечивает ее инвазию в двойную спираль с образованием D-петли
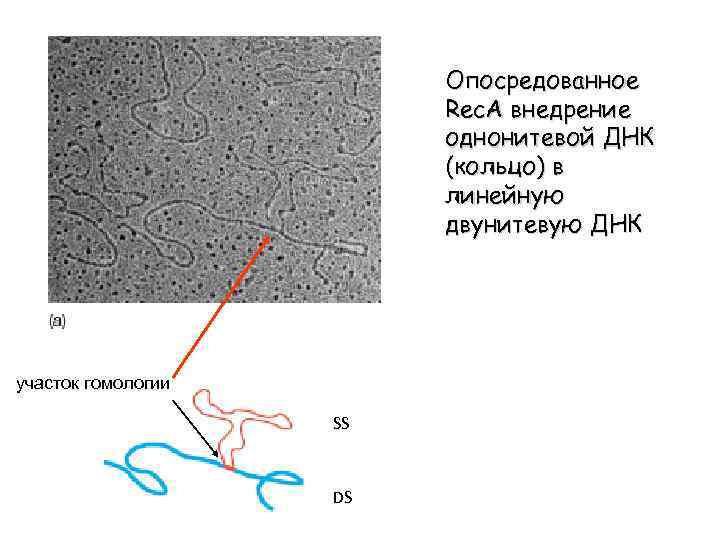
Опосредованное Rec. A внедрение однонитевой ДНК (кольцо) в линейную двунитевую ДНК участок гомологии SS 22. 13 DS Weaver, R. F. in Molecular Biology, 1999 Mc. Graw-Hill
Homologous Recombination Repairs ds Breaks В эукариотических клетках гомологом rec. A является Rad 51 Покрытая Rad 51 однонитевая ДНК внедряется в гомологичный участок сестринской хроматиды с образованием D петли 3’-конец внедрившейся цепи достраивается ДНК полимеразой и отжигается с 3’-концом комплементарной цепи исходного дуплекса Бреши застраиваются и однонитевые разрывы лигируются
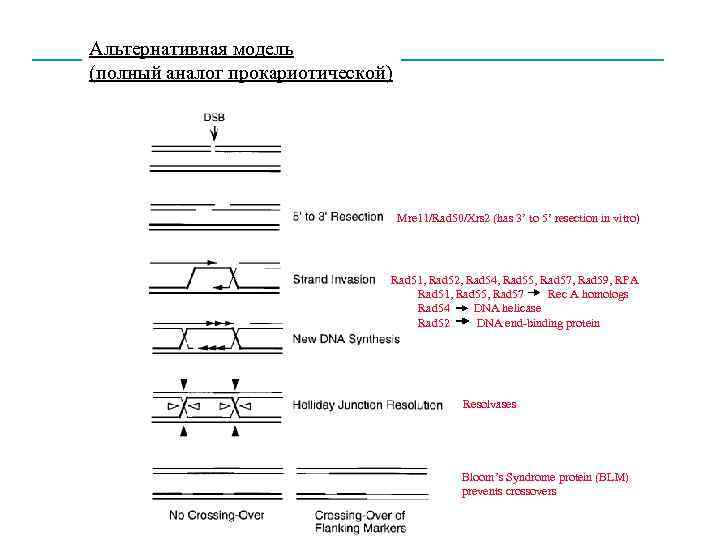
Альтернативная модель (полный аналог прокариотической) Mre 11/Rad 50/Xrs 2 (has 3’ to 5’ resection in vitro) Rad 51, Rad 52, Rad 54, Rad 55, Rad 57, Rad 59, RPA Rad 51, Rad 55, Rad 57 Rec A homologs Rad 54 DNA helicase Rad 52 DNA end-binding protein Resolvases Bloom’s Syndrome protein (BLM) prevents crossovers
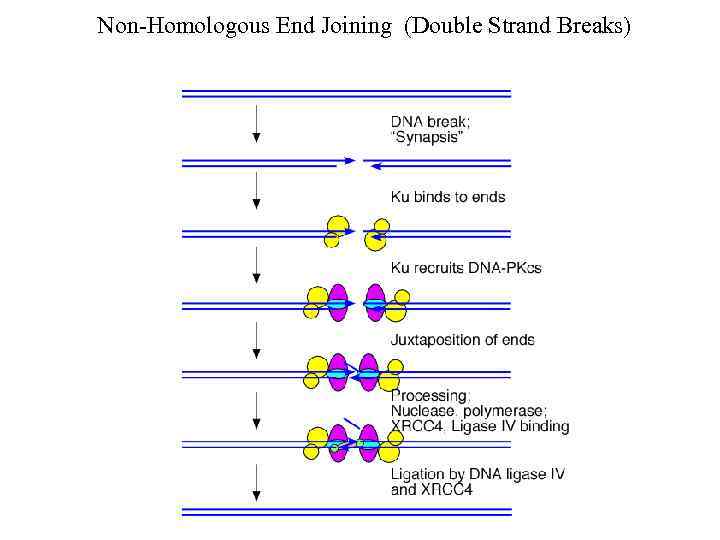
Non-Homologous End Joining (Double Strand Breaks)
РЕКОМБИНАЦИЯ ДНК Рекомбинация ДНК является одним из важнейших биологических процессов, повышающий уровень разнообразия геномов В рекомбинации ДНК участвуют многие механизмы, задействованные в репарации двунитевых разрывов по пути гомологической рекомбинации
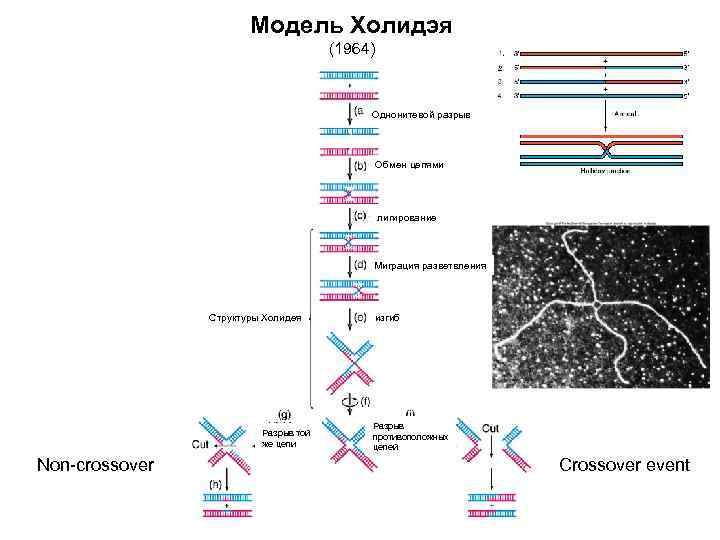
Модель Холидэя (1964) Однонитевой разрыв Обмен цепями лигирование Миграция разветвления Структуры Холидея Разрыв той же цепи Non-crossover изгиб Разрыв противоположных цепей Crossover event
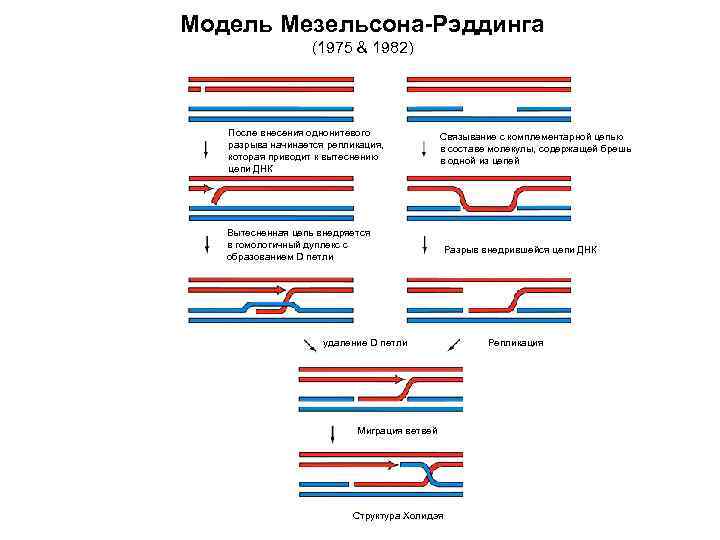
Модель Мезельсона-Рэддинга (1975 & 1982) После внесения однонитевого разрыва начинается репликация, которая приводит к вытеснению цепи ДНК Связывание с комплементарной цепью в составе молекулы, содержащей брешь в одной из цепей Вытесненная цепь внедряется в гомологичный дуплекс с образованием D петли удаление D петли Миграция ветвей Структура Холидэя Разрыв внедрившейся цепи ДНК Репликация
Рекомбинация в клетках E. coli Rec. BCD путь Rec. BCD комплекс вносит однонитевой разрыв рядом с 3’ концом сайта (5’-GCTGGTGG-3’) расплетает ДНК. сайты встречаются в среднем 1 раз на 5000 пн. Фрагмент однонитевой ДНК покрывается Rec. A белком, что обеспечивает его внедрение в гомологичный дуплекс В D петлю вносится разрыв (предположительно комплексом Rec. BCD) В результате происходит реципроктный обмен комплементарными цепями. После лигирования концов возникает типичная структура Холидэя Белки Ruv. A и Ruv. B (оба обладающие хеликазной и АТФазной активностями) обеспецивают миграцию ветвей. Ruv C внисит разрывы в структуру Холидэя, приводя к высвобождению двух молекул двунитевой ДНК
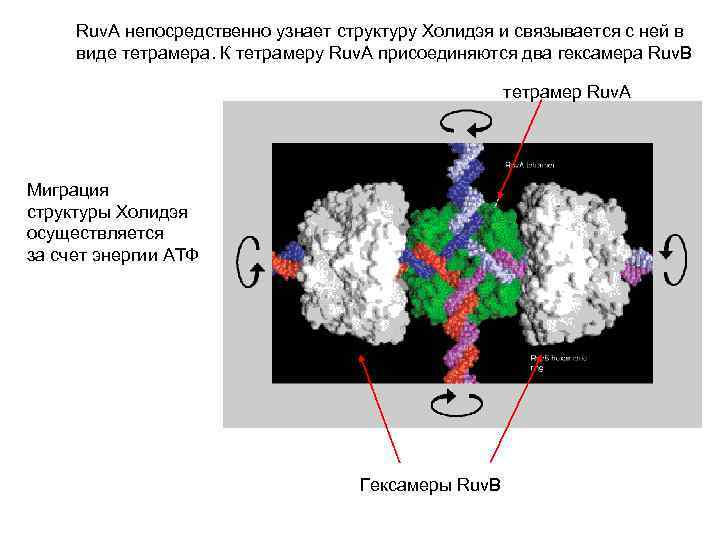
Ruv. A непосредственно узнает структуру Холидэя и связывается с ней в виде тетрамера. К тетрамеру Ruv. A присоединяются два гексамера Ruv. B тетрамер Ruv. A Миграция структуры Холидэя осуществляется за счет энергии АТФ Гексамеры Ruv. B
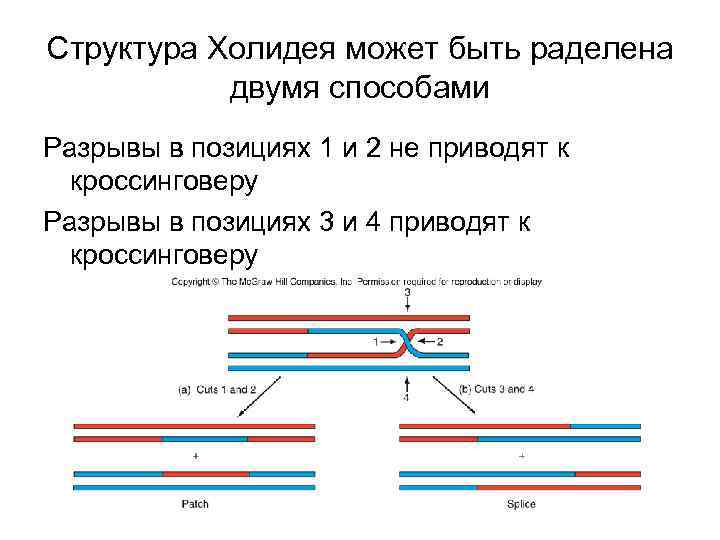
Структура Холидея может быть раделена двумя способами Разрывы в позициях 1 и 2 не приводят к кроссинговеру Разрывы в позициях 3 и 4 приводят к кроссинговеру
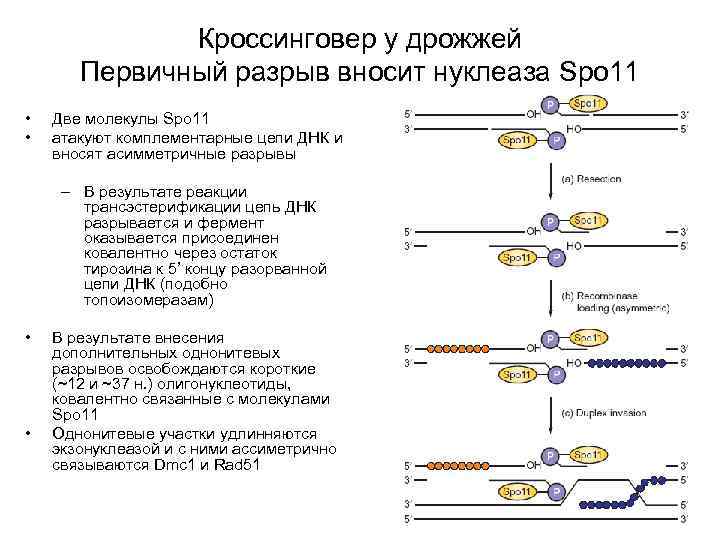
Кроссинговер у дрожжей Первичный разрыв вносит нуклеаза Spo 11 • • Две молекулы Spo 11 атакуют комплементарные цепи ДНК и вносят асимметричные разрывы – В результате реакции трансэстерификации цепь ДНК разрывается и фермент оказывается присоединен ковалентно через остаток тирозина к 5’ концу разорванной цепи ДНК (подобно топоизомеразам) • • В результате внесения дополнительных однонитевых разрывов освобождаются короткие (~12 и ~37 н. ) олигонуклеотиды, ковалентно связанные с молекулами Spo 11 Однонитевые участки удлинняются экзонуклеазой и с ними ассиметрично связываются Dmc 1 и Rad 51
Dmc 1 мейотический аналог Rad 51. Связывается с одним из однонитевых концов
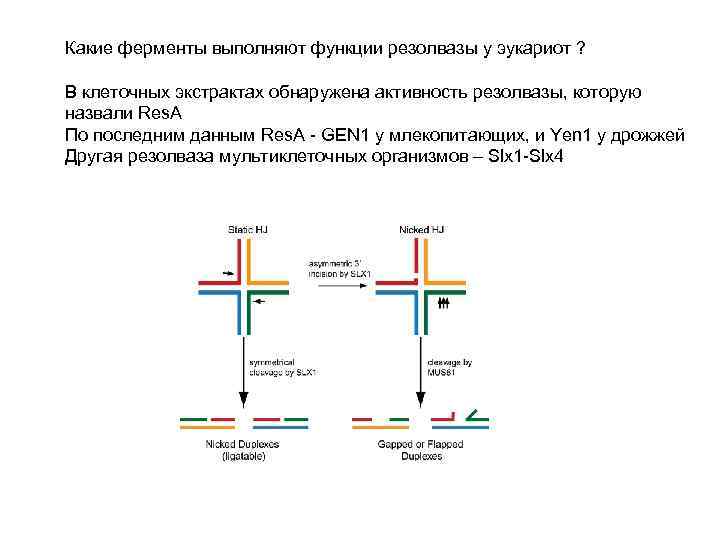
Какие ферменты выполняют функции резолвазы у эукариот ? В клеточных экстрактах обнаружена активность резолвазы, которую назвали Res. A По последним данным Res. A — GEN 1 у млекопитающих, и Yen 1 у дрожжей Другая резолваза мультиклеточных организмов – Slx 1 -Slx 4
ГЕННАЯ КОНВЕРСИЯ – «превращение» одного аллеля в другой
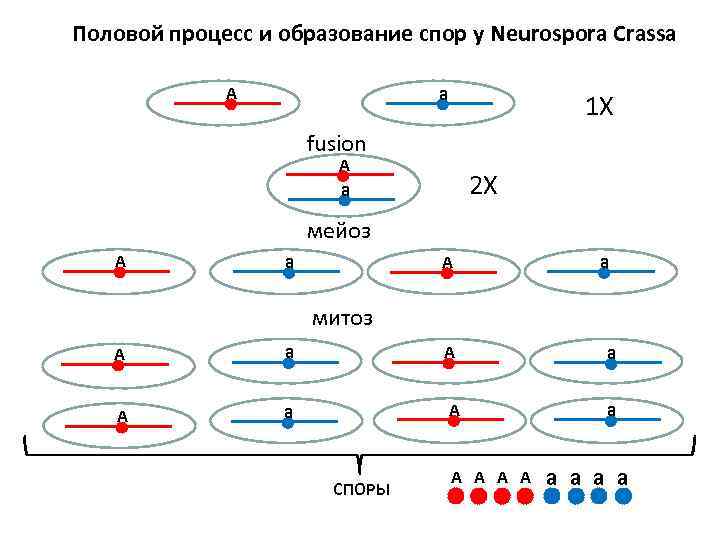
Половой процесс и образование спор у Neurospora Crassa а А 1 X fusion А 2 X а мейоз А а митоз А а А а СПОРЫ А А a a
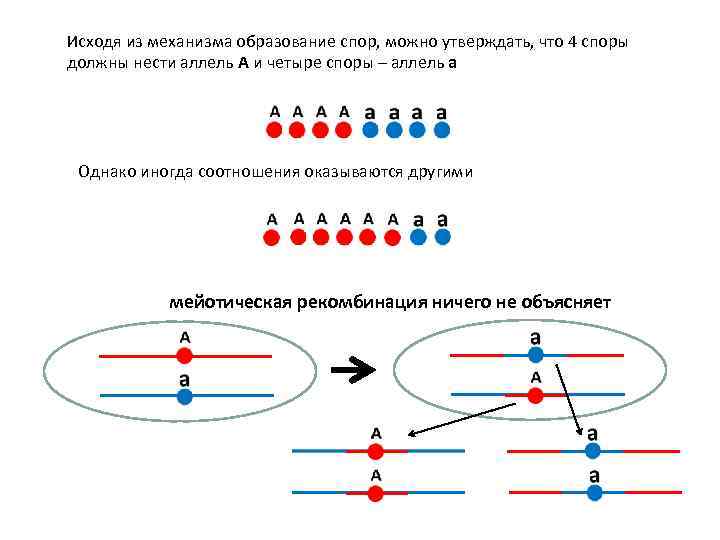
Исходя из механизма образование спор, можно утверждать, что 4 споры должны нести аллель A и четыре споры – аллель a Однако иногда соотношения оказываются другими мейотическая рекомбинация ничего не объясняет
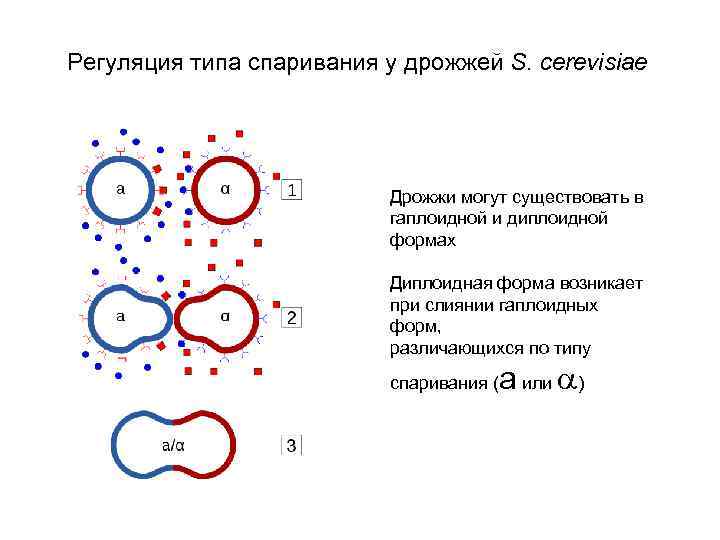
Регуляция типа спаривания у дрожжей S. cerevisiae Дрожжи могут существовать в гаплоидной и диплоидной формах Диплоидная форма возникает при слиянии гаплоидных форм, различающихся по типу спаривания ( a или a)
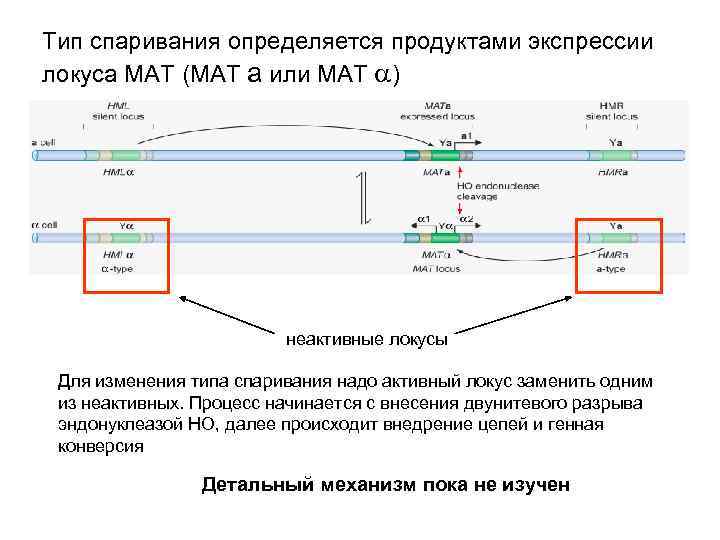
Тип спаривания определяется продуктами экспрессии локуса MAT (MAT a или MAT a) неактивные локусы Для изменения типа спаривания надо активный локус заменить одним из неактивных. Процесс начинается с внесения двунитевого разрыва эндонуклеазой HO, далее происходит внедрение цепей и генная конверсия Детальный механизм пока не изучен
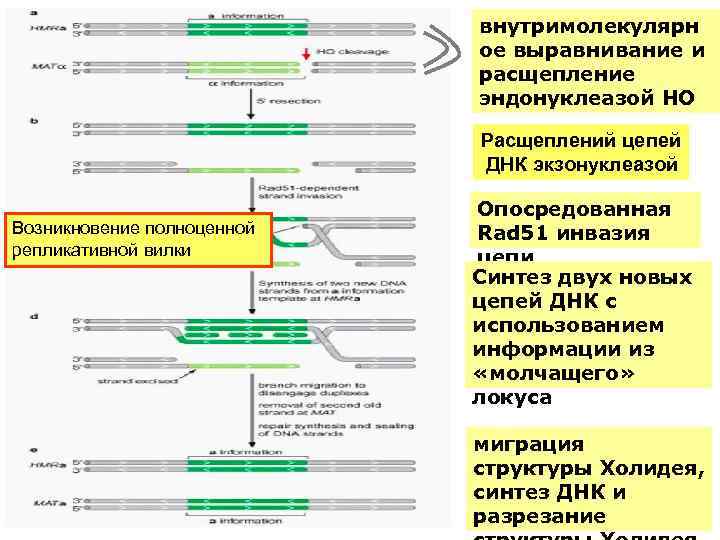
внутримолекулярн ое выравнивание и расщепление эндонуклеазой HO Расщеплений цепей ДНК экзонуклеазой Возникновение полноценной репликативной вилки Опосредованная Rad 51 инвазия цепи Синтез двух новых цепей ДНК c использованием информации из «молчащего» локуса миграция структуры Холидея, синтез ДНК и разрезание
Обмен участками может происходить и между сестринскими хроматидами
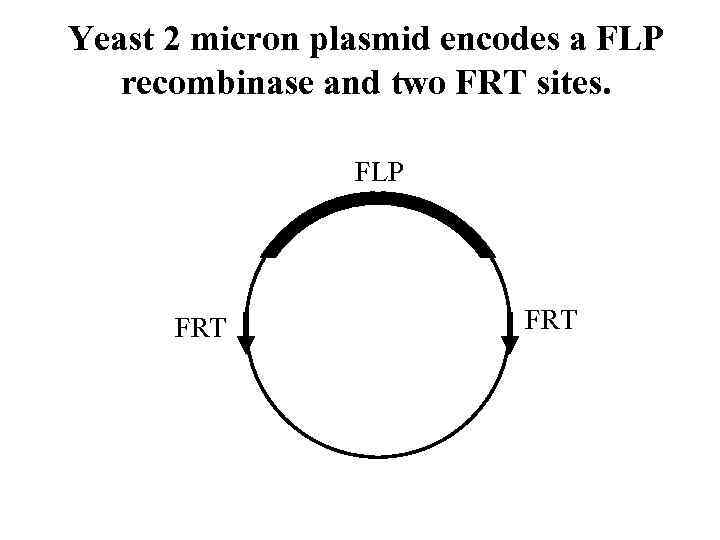
Yeast 2 micron plasmid encodes a FLP recombinase and two FRT sites. FLP FRT
FLP — Flippase FRT — Flippase Recognition Targets FRT – 34 bp последовательность ДНК с инвертированными 13 bp повторами на концах. FRT имеет направление благодаря асимметричному внутреннему участку 5′-GAAGTTCCTATTCtctagaaa. GTATAGGAACTTC-3′. 3’-CTTCAAGGATAAGagatcttt. CATATCCTTGAAG-5’ Флиппаза связывается с инвертированными повторами
в основном у прокариот сходство с топоизомеразами сохранение энергии при связывании белка с 3’ концом разорванной цепи ДНК Дальнейшие процессы идут без затраты энергии Альтернативная активация субъединиц связанных с «верхней» и «нижней» цепями ДНК исключает возможность появления двунитевых разрывов в молекуле ДНК
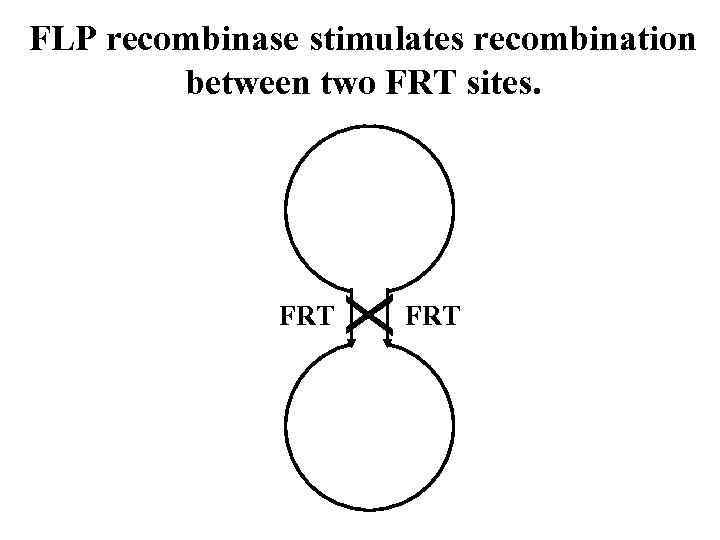
X FLP recombinase stimulates recombination between two FRT sites. FRT
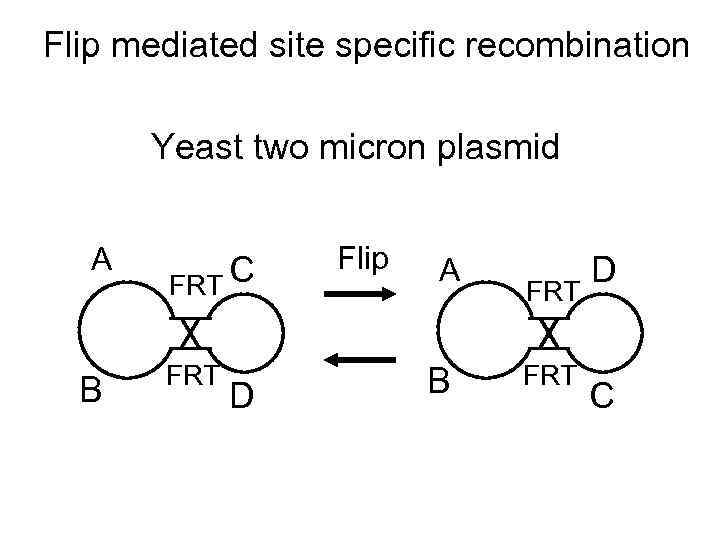
Flip mediated site specific recombination Yeast two micron plasmid A B FRT C FRT D Flip A B FRT D C
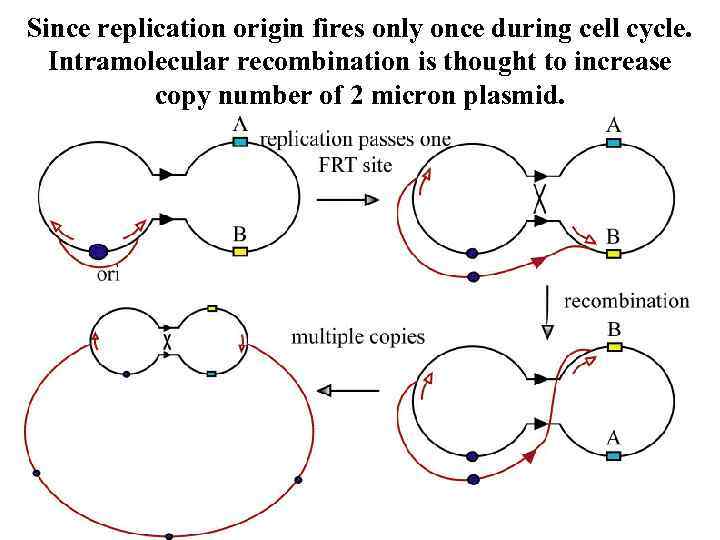
Since replication origin fires only once during cell cycle. Intramolecular recombination is thought to increase copy number of 2 micron plasmid.
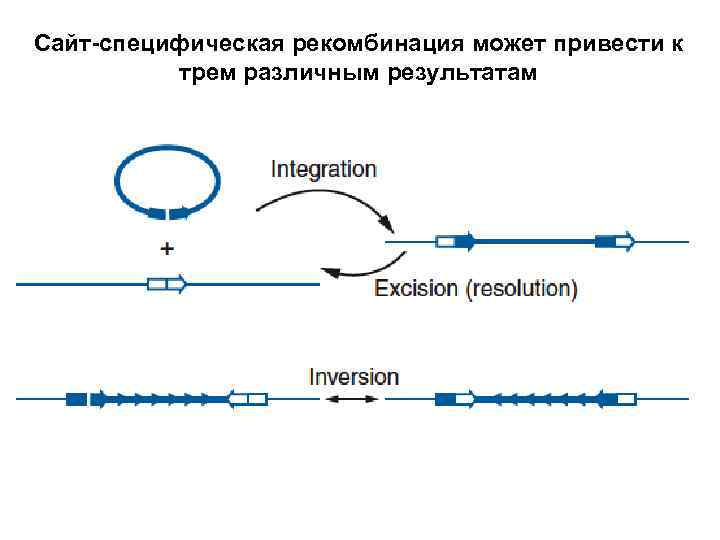
Сайт-специфическая рекомбинация может привести к трем различным результатам
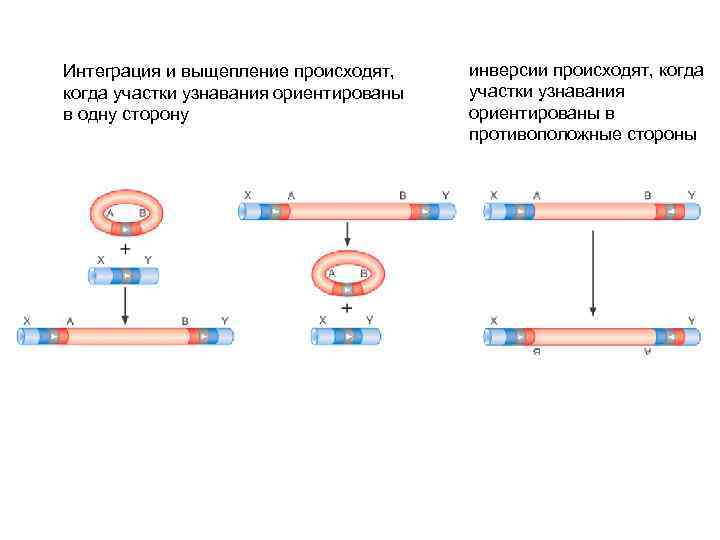
Интеграция и выщепление происходят, когда участки узнавания ориентированы в одну сторону инверсии происходят, когда участки узнавания ориентированы в противоположные стороны
В ходе работы рекомбиназ возникает временны промежуточный комплекс рекомбиназы с разорванной цепью ДНК, который запасает энергию, необходимую для последующего соединения цепей Существует два класса сайт-специфических рекомбиназ, различающихся по аминокислотному остатку активного центра, участвующему в образовании ковалентной связи с ДНК: тирозиновые и сериновые
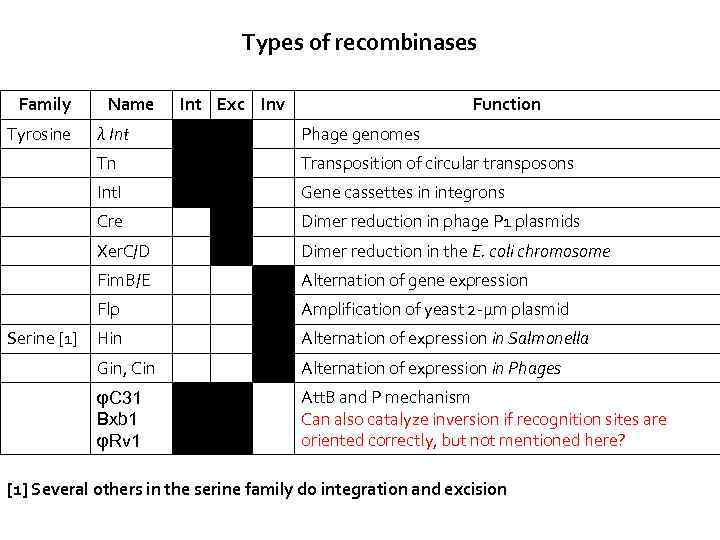
Types of recombinases Family Tyrosine Name Int Exc Inv Function Phage genomes Tn Transposition of circular transposons Int. I Gene cassettes in integrons Cre Dimer reduction in phage P 1 plasmids Xer. C/D Dimer reduction in the E. coli chromosome Fim. B/E Alternation of gene expression Flp Amplification of yeast 2 -μm plasmid Hin Alternation of expression in Salmonella Gin, Cin Alternation of expression in Phages φC 31 Bxb 1 φRv 1 Serine [1] λ Int Att. B and P mechanism Can also catalyze inversion if recognition sites are oriented correctly, but not mentioned here? [1] Several others in the serine family do integration and excision
Обновлено 23.09.2022

Описание ситуации
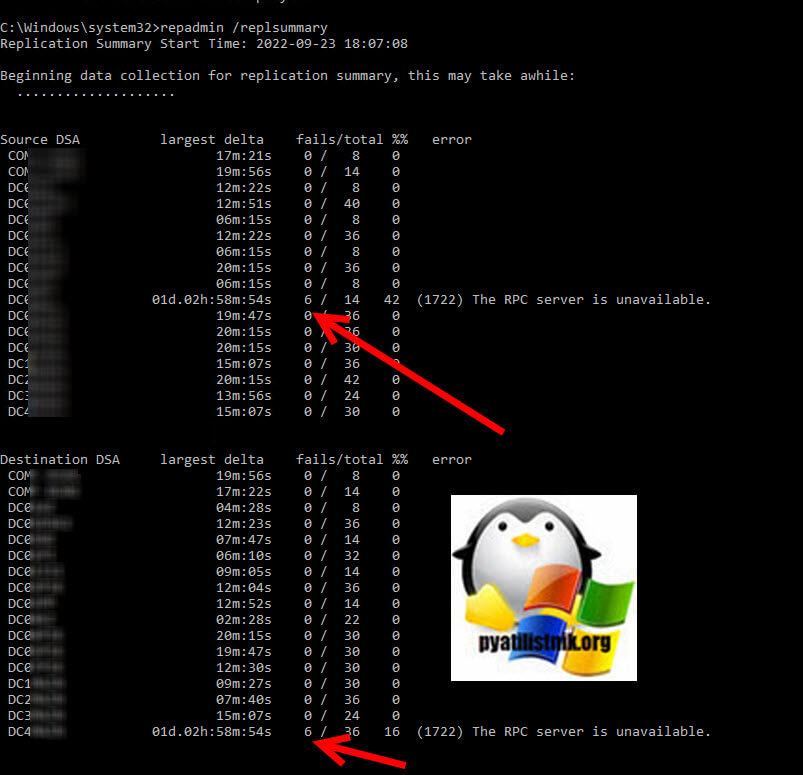
И так у меня есть лес и 3 домена Active Directory, развитая сеть сайтов AD. Утром в систему мониторинга поступило уведомление, что появились проблемы с репликацией между контроллерами домена, при выводе команды repadmin /replsummary, я увидел ряд ошибок:
- (8524) The DSA operation is unable to proceed because of a DNS lookup failure
- (1722) The RPC server is unavailable.
Если с ошибкой «(1722) The RPC server is unavailable.» я сталкивался и ее успешно решал, то вот «(8524) The DSA operation is unable to proceed because of a DNS lookup failure» я видел впервые.
Также я вам советую сразу запустить массовую диагностику и выгрузить весь результат в текстовые файлы, для этого создайте bat-файл вот с таким содержанием:
@echo off
chcp 855
repadmin /replsummary > c:tempreplsummary.log
repadmin /syncall > c:tempsyncall.txt
repadmin /Queue > c:tempQueue.txt
repadmin /istg * /verbose > c:tempistg.txt
repadmin /bridgeheads * /verbose > c:tempbridgeheads.txt
repadmin /syncall /APed > c:tempAPed.txt
dcdiag /a /q > c:tempdcdiag.txt
repadmin /showrepl > c:tempshowrepl.txt
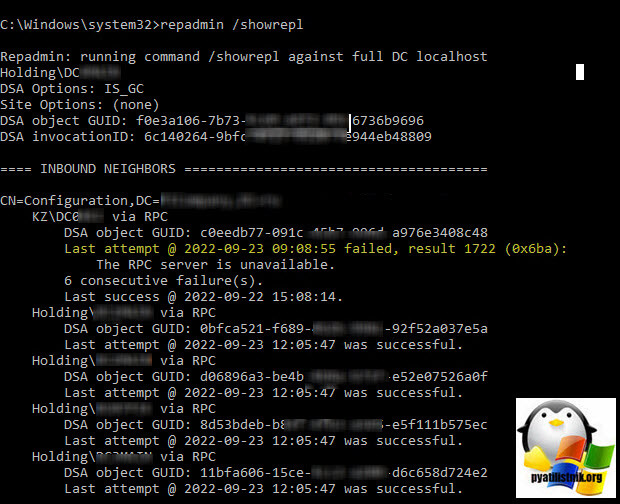
Например я также через showrepl вижу ошибку:
Last attempt @ 2022-09-23 09:09:37 failed, result 1722 (0x6ba):
Как исправить ошибку репликации (8524)
Первое, что вы должны выполнить, это убедиться:
- 1️⃣Что ваши контроллеры домена доступны по сети, вы точно должны увидеть, что порты по которым работает активный каталог, нормально доступны с контроллеров домена на которых нет проблем. Самое важное, чтобы все DNS-имена нормально резолвелись. Очень часто работающие контроллеры ссылаются на состояние 8524, если контроллер домена Active Directory не может разрешить удаленный DC с помощью полностью CNAME-записи.
- 2️⃣Для проверки портов я советую использовать утилиту Telnet или PowerShell
- 3️⃣100% быть уверенным, что на сбойном контроллере нет ошибки и состояния (1722) The RPC server is unavailable
- 4️⃣Если с портами и доступностью все хорошо, и все контроллеры друг друга видят без ошибок, то попробуйте выполнить принудительную реплику по всем контроллерам через команду (Лучше это делать сначала на любом рабочем DC, потом можно на сбойном или имеющем ошибки):
Данная команда выполнятся может долго, зависит от количества сайтов и контроллеров домена.
- 5️⃣Проверьте правильность настройки ваших сайтов Active Directory, нет ли там устаревших связей, или сайтов которых уже нет, в которых могут быть уже выведенные контроллеры домена. Если такие присутствуют, то удалите сайт и такие объекты. На проблемы с сайтами может указывать ошибка с ID 8524, чтобы решить эту проблему, запустите сайты и службы Active Directory (dssite.msc). Затем перейдите в раздел «Серверы», выберите удаленный контроллер домена — Настройки NTDS и удалите подключения, удалите настройки NTDS, удалите от туда контроллер домена
- 6️⃣Обязательно проверьте в просмотре событий журналы «СИСТЕМА«, «Active Directory Web Services«, «DFS Replication«, «Directory Service«, «DNS Server» наличие альтернативных ошибок, которые вам могут, что-то подсказать, куда дальше копать.
Тут вы можете обнаружить ряд событий, которые будут причиной ошибки репликации «(8524) The DSA operation is unable to proceed because of a DNS lookup failure»:
-
- ID 1926 — Попытка установить ссылку репликации на раздел каталога только для чтения со следующими параметрами не удалась
- ID 1925 — Попытка установить ссылку на репликацию для следующего раздела каталога writable не удалась.
- ID 1865 — KCC не смог сформировать полную топологию сети деревьев. В результате, следующий список сайтов не может быть достигнут с локального сайта
- ID 1308 — KCC обнаружил, что последовательные попытки реплицироваться со следующей службой каталогов последовательно сбой.
- ID 1655 — Active Directory попытался связаться со следующим глобальным каталогом, и попытки оказались безуспешными
- ID 2023 — Этот сервер каталогов не смог реплицировать изменения на следующий удаленный сервер каталога для следующего раздела каталога
- 7️⃣Если сбойный контроллер домена уже не будет в сети и просто не может работать, то нужно его полностью удалить с очисткой метаданных.
- 8️⃣Можете еще попробовать поискать дополнительные подсказки через инструмент Active Directory Replication Status Tool
После того, как сбойный контроллер домена был введен в боевое состояние, проверены нужные пункты из указанных выше, реплика восстановилась. Об этом стало говорить уменьшающееся количество ошибок при попытке реплики.
Надеюсь, что смог чем-то помочь, с вами был Иван Семин, автор и создатель IT портала Pyatilistnik.org.
Мутагены — факторы окружающей среды, которые способны вызывать ошибки при нормальной репликации ДНК, что ведет к спонтанным мутациям. Ошибки в репликации ДНК зависят от температурных условий, pH, состава среды. Особенно мощными мутагенными факторами являются ультрафиолетовые и ионизирующие излучения. К мутагенам химической природы относятся аналоги азотистых оснований, отдельные красители акридинового ряда, алкилирующие соединения, некоторж антибиотики, гидроксиламин, уретан и азотистая кислота. [c.61]
Химическое изменение оснований. Некоторые мутагенные вещества действуют путем химического изменения содержащихся в ДНК оснований, что приводит к ошибкам репликации. Вполне понятное изменение вызывает нитрит. Азотистая кислота дезаминирует аденин, гуанин или цитозин без разрыва или каких-либо других изменений полинуклеотидной цепи. В результате замещения аминогруппы гидроксильной группой аденин превращается в гипоксантин и спаривается с цитозином вместо тимина, что приводит к мутации АТ СС. Если цитозин дезаминируется в урацил, то он спаривается с аденином вместо гуанина, и это ведет к мутации СС -АТ. Будучи превращен в ксантин, гуанин по-прежнему спаривается с цитозином, т. е. дезаминирование С не вызывает мутации. Гидроксиламин вступает в реакцию главным образом с цитозином и изменяет его так, что тот спаривается с аденином значит, он тоже вызывает мутации СС ТА. [c.444]
Ошибки репликации ДНК приводят к мутациям [12] [c.128]
Мутации как ошибки репликации ДНК. Данные, полученные на человеке, свидетельствуют о существовании тесной связи между мутациями и клеточными делениями. Импульсом к изучению этой проблемы послужила гипотеза о механизме возникновения точковых мутаций Уотсона и Крика (рис. 5.26) [1347]. Важную роль в этом сыграли и ранние исследования на микроорганизмах, из которых следовало, что многие спонтанные мутации действительно возникают во время репликации ДНК в результате ошибочной вставки неправильного нуклеотида, приводящей к появлению в будущих клеточных поколениях новой, отличающейся пары оснований. В последние десятилетия получено до удивления мало новых данных о механизмах возникновения спонтанных мутаций, что очень сильно контрастирует с громадным объемом имеющихся в настоящее [c.191]
Заметим, что различие по длине рестрикционных фрагментов между родителями и детьми может возникать не только в результате точковых мутаций в сайте узнавания, но и в результате ошибки репликации или кроссинговера. Ожидается, что эти события происходят чаще, чем точковые мутации. Среди 27 индивидов, у которых проанализировано 240 полос, выявлена такая полоса, которой не было ни у одного из родителей, отсюда скорость мутирования составляет 1/240, что по крайней мере на 4 порядка выше, чем скорость мутирования для точковых мутаций (разд. 2.3.3.9). [c.290]
Передача наследственной информации в неискаженном виде — важнейшее условие выживания как каждого конкретного организма, так и вида в целом. Следовательно, в ходе эволюции должна была сформироваться система, позволяющая клетке исправлять нарушения ДНК, вызванные ошибками репликации или повреждающими воздействиями окружающей среды. Подсчитано, что в результате повреждений, обусловленных этими причинами, в геноме клеток зародышевой линии человека происходит в среднем шесть нуклеотидных замен в год. По-видимому, в соматических клетках за год происходит примерно такое же число мутаций. [c.79]
В работе [59] было показано, что 95% мутаций, индуцируемых АП в Е.соЫ, относятся к транзициям А—Т — -> Г — Ц, т. е. преимущественно возникают ошибки репликации. [c.34]
Данные таблицы указывают, что в Si состоянии А и Т (У) легче, а Ц и Г труднее переходят в редкие формы, чем в состоянии Sq. Видно также, что в случае амино-иминной таутомерии А более склонен к таутомеризации, чем Ц лактам-лактимные превращения Т (У) более вероятны, чем Г. Таким образом, в состоянии Si положение противоположно тому, что имеется в So- Поэтому при действии УФ-света ошибки репликации чаще всего приводят к транзициям А—Т Г—Ц (рис. 3, а и б), а ошибки включения — к транзициям Г —Ц- А—Т (рис. 3, ж и з). Рассмотренный механизм объясняет возникновение мутаций, отнесенных к транзициям А — Т — Г — Ц [177]. [c.54]
ДНК-полимеразы проверяют комплементарность каждого нуклеотида матрице дважды один раз перед включением его в состав растущей цепи и второй раз перед тем, как включить следующий нуклеотид. Очередная фосфодиэфирная связь образуется лишь в том случае, если последний (З -концевой) нуклеотид затравки комплементарен матрице. Если же на предыдущей стадии полимеризации произошла ошибка (например, из-за того, что нуклеотид в момент полимеризации находился в необычной таутомерной форме), то репликация останавливается до тех пор, пока неправильный нуклеотид не будет удален. Некоторые ДНК-полимеразы обладают не только полимеризующей, но и 3 -экзонуклеазной активностью, «Которая отщепляет не спаренный с матрицей нуклеотид затравки. После чего полимеризация восстанавливается, от механизм, коррекция, заметно увеличивает точность работы ДНК-полимераз. Мутации, нарушающие З -экзонуклеазную активность ДНК-полимеразы, существенно повышают частоту возникновения прочих мутаций. Напротив, мутации, приводящие к усилению экзонуклеазной актив- ности относительно полимеризующей, снижают темп мутирования Генетического материала. [c.47]
Спонтанные генные мутации определяются ошибками при репликации ДНК, возникающими вследствие теплового движе-иия атомов и молекул. Очевидно, что ошибки транскрипции и трансляции не наследуются. [c.283]
Если ошибка синтеза не устраняется системами репарации, то неизбежна деформация дуплекса и искажение генетической программы. Такие сохраняющиеся при репликации изменения ДНК носят название мутации. Они могут быть спонтанными и индуцированными. Частота спонтанных мутаций невелика и составляет всего 10 —10 на клетку. В основном имеют место мутации, обусловленные действием внешних факторов физических (радиация), биологических (вирусы) и чужеродных химических веществ на генетический аппарат клеток. Наиболее многочисленными и опасными являются мутагены окружающей среды. Загрязнение воды и воздуха различными химическими отходами промышленных предприятий, химическими средствами защиты растений отрицательно сказывается на генетической программе всех живых организмов. В последние годы установлено, что ряд пищевых красителей, стабилизаторов и вкусовых добавок обладает выраженной мутагенной активностью, что привело к значительному ужесточению требований, связанных с применением химических веществ в пищевой промышленности. Многие лекарственные вещества также воздействуют на генетический аппарат клеток и должны подвергаться специальным генетическим испытаниям. [c.455]
Из уровня спонтанных мутаций у бактерий в расчете на одно поколение рассчитано, что вероятность одной репликационной ошибки при синтезе ДНК составляет порядка 10 . Эту величину можно рассматривать как отношение скоростей реакций правильной репликации [c.194]
Как обсуждалось в гл. 13, наследственная информация, заключенная в нуклеотидной последовательности ДНК, сохраняется неизменной благодаря действию сложных метаболических механизмов, обеспечивающих осуществление репликации и репарации. Мутации могут быть результатом ошибки на любом из многочисленных последовательных этапов этих процессов. Мутагенные факторы способны изменять как непосредственно структуру ДНК, так и структуру ферментов, прямо или косвенно участвующих в соответствующих метаболических процессах. Для понимания механизмов мутаций требуется знание нуклеотидной последовательности гена дикого типа и мутантного гена. Без этого невозможно понять связь между изменениями, происходящими в структуре ДНК и действием конкретных факторов или условий среды, вызывающих мутации. Современные методы клонирования генов сделали возможным прямое определение нуклеотидной последовательности ДНК. Однако еще совсем недавно при изучении молекулярной природы мутаций приходилось анализировать аминокислотные замены в белках, синтезируемых мутантными генами, а затем с помощью таблиц генетического кода выявлять изменения в нуклеотидной последовательности. [c.8]
Связь между ДНК, менделевскими факторами и признаками организма не столь проста, как представлялось прежде, но под наблюдения классических менделистов можно тем не менее подвести химическую основу. Установленные ранее явления доминантности и рецессивности рассматриваются теперь в таких аспектах, что можно без труда показать связь этих явлений с определенными химическими реакциями (разд. 2.1.2). Эпистаз (разд. 2.1.2) можно интерпретировать в понятиях взаимодействующих генов и генных продуктов. Нетрудно, например, представить себе, как может происходить химическое воздействие на экспрессию на уровне как самого гена, так и любого из ряда последовательных генных продуктов химическими факторами, содержащимися во внутренней и во внешней среде. Наконец, мутацию можно интерпретировать как ошибку в репликации ДНК. Такая ошибка может ограничиваться замещением одного нуклеотида другим (точковая мутация) или же выражаться в структурной перестройке целых хромосом (хромосомные мутации, в том числе нехватки, удвоения, транслокации и инверсии). [c.38]
Если УФ-свет вызывает мутацию гена, ответственного за биосинтез ДНК-полимеразы или других ферментов репарирующей системы, то возникшие мутантные клетки характеризуются высокой частотой спонтанных мутаций, которые не связаны с действием внешних или внутренних мутагенных факторов, а обусловлены ошибками в ходе репликации. В связи с этим в генетике возникло представление о гене, контролирующем частоту естественных мутаций,— гене-мута-торе. [c.311]
Устойчивость процесса отбора по отношению к систематическим помехам зависит прежде всего от уровня помех, т. е. от вероятности мутаций в единицу времени и вероятности ошибочной репликации. Следует учитывать также различие в характере повреждений, вызываемых помехами. При ошибочной редупликации (или в результате мутаций) могут возникнуть как летальные варианты, т. е. гиперциклы с весьма малой репродуктивной способностью, так и гиперциклы, способные к дальнейшему развитию, но отличающиеся от исходного. Характер повреждений зависит от стадии развития. На первой стадии (до возникновения адаптеров) все мутации и ошибки ведут к равноправным вариантам, летальных вариантов нет. На второй стадии, после образования адаптеров, но до возникновения единого кода летальные мутации существенной роли не играют, поскольку сводятся к уменьшению скорости репродукции. [c.36]
На наших глазах очередной кризис возник и в молекулярной генетике. Совсем недавно доминировали представления о необычайной точности и стабильности механизмов репликации и процессинга РНК, в результате чего мутации (ошибки репликации) возникают крайне редко, с частотой порядка 10 10 за одно поколение. Немало приводилось убедительных доводов в пользу того, что только такая надежность репликации ДНК и обеспечивает саму возможность прогрессивной эволюции — при учащении мутационного процесса отбор-де не справился бы с задачей поддержицання упорядоченности организации и совершевствовавяя [c.4]
По происхождению мутации делятся на спонтанные (неконтролируемые) и индуцированные (контролируемые). Первые возникают в результате неконтролируемого влияния каких-то естественных факторов (радиация, температура и т. д.). Направленное использование мутагенов приводит к возникновению индуцированных мутаций. Многими экспериментами четко показано, что мутации возникают независимо от условий среды обитания, т. е. не направленно. Мутации возникают в основном как ошибки репликации ДНК. Выделяют следующие типы мутаций перестройка хромосом, перестройка генома клетки грибов и водорослей (полиплоидия, гаплоидия, гетероплоидия), внутригенные изменения (прямые мутации, реверсии, обратные мутации). [c.102]
В качестве альтернативного подхода к измерению скоростей мутации А. Portner и соавт. [198] использовали моноклональные антитела и сравнили скорости мутации в отношении к резистентности антител к вирусам гриппа, везикулярного стоматита и Синдбис. Для всех трех вирусов скорости мутации составили около на репликацию, тогда как скорость ошибки репликации ДНК Е. соИ оценивалась как 10 —10 ° [63]. [c.190]
Спонтанные мутации возникают случайно, т.е. в любой. момент любой ген может претерпеть изменения. Причинами спонтанного мутационного процесса являются многочисленные факторы экзогенной и эндогенной природы, в том числе постоянное воздействие па организм человека мутагенов химической, биологической и физической природы (например, естественный фон облучения, действие вирусов) ошибки репликации ДНК, которые копируются и накапливаются в ряду клеточных поколений нарушение функционирования репаративных систем действие экзогенных метаболитов физи0 ю1ическ0е состояние и возраст организма. Спонтанные мутации могут возникать как в половых, так и в соматических клетках на генном, хромосомном и геномном уровнях. [c.125]
Разные аллели одного и того же Г. возникают благодаря мутациям-илслецуемьш изменениям в структуре исходного Г. В норме Г. чрезвычайно стабилен и при удвоении хромосом во время репликации ДНК воспроизводится совершенно точно вероятность ошибки не превышает 10″ . Мутации происходят редко и обычно влекут за собой неблагоприятные последствия для организма, т. к. нарушается его способность синтезировать нормальный белок. Однако в целом это явление играет положит, роль накопление редких полезных мутаций создает основу генетич. изменчивости, необходимой для эволюции. [c.517]
Равновесие между созидательными возможностями выбора среди специфических оснований в ДНК (созидательные мутации) и точностью синтеза белков (поддерживающих жизнь организма) является основой эволюции. Ферменты, которые заряжают тРНК специфической аминокислотой, обладают очень низкой вероятностью ошибки, порядка 1 Ю» для гомологичных аминокислот. При репликации точность даже выше, и величина ошибки редко превышает 1 на 10. [c.212]
Приближенная модель репликации ДНКизображена на рис. 2.11. Из приведенной схемы видно, что репликация точно воспроизводит прежнюю (исходную) структуру ДНК. Но если произошла ошибка в процессе копирования (мутация), то она будет с предельной точностью копироваться при последующих репликациях изменившейся ДНК. Показано, что участки ДНК, содержащие скопления нуклеотидов, обладают повышенной склонностью к спонтанным мутациям [22]. [c.94]
Для нормального функционирования аппарата исправления ошибок, связанных с включением неправильных нуклеотидов, необходимо располагать механизмом, позволяющим отличать новосинтезированную цепь ДНК от родительской матричной цепи. В противном случае с вероятностью 1/2 будет происходить исправление нуклеотида в родительской цепи, приводящее к закреплению потенциально мутагенной ошибки, допущенной ДНК-полимеразой. Вероятно, для установления различий между родительской и дочерней цепями ДНК в Е. соИ используется метилирование аденина в последовательности GAT . Эта палиндром-ная последовательность обычно метилирована в обеих цепях родительской ДНК. При полуконсервативной репликации метилированной ДНК образуется дочерняя ДНК, в которой одна цепь, пришедшая от родительской ДНК, метилирована, а новообразованная цепь в течение некоторого времени после выхода из области репликативной вилки остается неметилированной. Следует заметить, что метилирование новообразованной цепи ДНК осуществляется ферментом, отличным от метилаз, входящих в систему рестрикции—модификации, обсуждавшуюся в гл. 9. Бактерии dam , дефектные по метилированию аденина в результате нарушения синтеза соответствующей метилазы характеризуются повышенной частотой спонтанных мутаций, что подтверждает гипотезу об участии метилазы dam в системе исправления ошибок репликации. [c.123]
Анализ приведенных выше результатов дает возможность написать для преобладающих таутомерных форм оснований нуклеиновых кислот формулы, изображенные на фиг. 55. Минорные таутомерные формы, возможно, играют существенную роль в возникновении спонтанных мутаций, поскольку спаривание несоответствующих оснований (см. гл. ХУП1) должно привести к ошибке при включении оснований и при последующей репликации цепи. Можно показать, что если скорость включения основания в цепь нуклеиновой кислоты меньше скорости перехода минорного таутомера в доминирующую форму, то скорость спонтанных мутаций, обусловленных данным основанием, приблизительно равна константе равновесия между минорным и доминирующим таутомерами. К сожалению, для азо- [c.308]
Задание 189. Напишите программу для моделирования самоорганизации ДНК в качестве примера самоорганизуюшихся систем. Используйте для этого следующую простую модель. Пусть имеется 100 молекул ДНК, состоящих из 12 нуклеотидов четырех видов (их обозначим буквами А, Т, С и G). Последовательность нуклеотидов в этих 100 молекулах ДНК случайная. Назовем одну из последовательностей идеальной она имеет некоторые преимущества перед остальными. Из 100 молекул ДНК в результате репликации получается еще 100 молекул. Однако при репликации встречаются ошибки (мутации), например в количестве 1%. Теперь из 200 молекул 100 погибает. При этом имеет значение преимущество, которым обладают молекулы с последовательностью нуклеотидов, похожей на идеальную . (Например, при каждом совпадении нуклеотида и его положения в цепи с идеальной последовательностью вероятность гибели уменьшается в два раза.) Процессы репликации и гибели протекают очень быстро. В конце концов все молекулы ДНК должны получить идеальную последовательность нуклеотидов, хотя вероятность ее образования в результате случайного процесса составляет 1 16777216. Что будет, если мутации будут возникать чаще или реже [c.330]
Теперь мы уже вполне подготовлены к тому, чтобы приступить к вопросу, поставленному в гл. VU, а именно к вопросу о молекулярном механизме возникновения тех изменений в последовательности нуклеотидов ДНК, которые приводят к мутациям. Действительно, исследование характера возникновения мутаций Т-четных фагов с использованием методов генетического анализа с высоким разрешением дает большие возможности для проникновения в природу мутационного процесса. Использование фагов имеет еще одно важное преимущество по сравнению с ис-лользованием бактерий. Мутации фаговой ДНК можно изучать как в том случае, когда она находится в состоянии покоя вне клетки в составе инфекционной фаговой частицы, так и когда она находится в реплицирующемся, внутриклеточном, вегетативном состоянии. Уже самые первые исследования Херши и Лурия показали, что частота спонтанных мутаций в покоящейся ДНК очень мала — столь мала, что в течение многих лет считалось (как потом оказалось, ошибочно), что внеклеточные фаговые частицы вообще не мутируют месяцами и даже годами. Таким образом, новые мутации появляются в основном во время вегетативного размножения фага в клетке-хозяине. Рассмотрим следующий пример. Культуру Е. oli заражают препаратом фага Т2/- с титром 10 частица/мл. Фагу дают размножиться в течение нескольких циклов, пока все бактерии в культуре не подвергнутся лизису, а титр фага не достигнет величины 10 частица/мл. Оказывается при этом, что с каждым циклом размножения доля г-мутантов во всей популяции фагов увеличивается (примерно с 10″ в начале до 10 в конце). Следовательно, мутанты фага возникают в результате ошибок копирования при внутриклеточной репликации его генетического материала. Репликация ДНК родительского фага является очень точным процессом. И все же при репликации иногда происходит ошибка, порождающая в одной из вегетативных реплик изменение последовательности нуклеотидов, или мутацию. Мутантная реплика генетического материала включается затем при созревании в инфекционную фаговую частицу, которая в свою очередь заражает новую бактериальную клетку. В этой клетке очень точно копируется уже измененная информация, содержащаяся в мутантной частице поэтому все потомство такой частицы оказывается тоже мутантным. Поскольку репликация ДНК вегетативного фага происходит в соответствии с постулированным Уотсоном и Криком полуконсервативным механизмом, размножение фагового генома можно рассматривать как процесс бинарного деления и с точки зрения статистического анализа совершенно аналогичным процессу размножения генома бактерий. Следовательно, уравнение, связывающее долю мутантных особей п среди общего числа N потомков одного исходного родителя, возникших после g генераций, с частотой мутаций а [c.315]
Если элементарное мутационное событие представляет собой [включение неправильного нуклеотида в определенный участок синтезируе-мой полинуклеотидной реплики и если ДНК вегетативного фага реплицируется в соответствии с полуконсервативным механизмом Уотсона и Крика, то мы можем предсказать такую особенность вновь рождаюш егося мутантного генома, которую без знания молекулярной основы процесса мутирования вообще невозможно было бы предвидеть. Предположим, что во время синтеза цепи-реплики происходит одна из редких ошибок копирования, например остаток тимина в родительской цепи незаконно спаривается с гуанином, а не с аденином. В результате этого мутагенного акта репликации возникает двойная спираль с исходной ин-формацией в старой (родительской) цепи и мутантной информацией в цепи, синтезированной заново (фиг. 160). При следующем цикле репликации комплементарные нити этой мутантной молекулы вновь разъединяются и каждая из них, функционируя как матрица, синтезирует новую комплементарную цепь. В результате появляется одна двойная спираль ДНК, несущая мутантную информацию в обеих цепях, и одна немутантная двойная спираль. Исходная мутантная молекула ДНК представляет собой, следовательно, гетеродуплексную гетерозиготу, которая несет в одном участке два аллеля — мутантный и немутантный, по которым при следующем цикле репликации происходит расщепление. Можно ожидать, что во время внутриклеточного размножения фага некоторые молекулы ДНК фага с мутацией, возникшей в результате ошибки копирования при последней репликации, будут извлечены из вегетативного фонда фага и войдут в состав зрелых инфекционных частиц. Эти частицы и будут мутационными гетерозиготами. [c.325]
Во-первых, исходным повреждением, отвечающим за потенциальный мутагенный эффект, должны быть тиминовые димеры. Во-вторых, процессом, превращающим потенциальные изменения в окончательные мутации, не может быть репарация за счет иссечения и заполнения (она могла бы приводить к мутации, если бы, например, точность репарационной репликации, изображенной на фиг. 187, была невелика и допускала ошибки копирования). Можно заключить, следовательно, что мутация вызывается нерепарированными тиминовыми димерами в тех клетках, которые выжили, несмотря на наличие такого неисправленного п овреж- [c.382]
Другой путь возникновения транзиций-это случаи ошибочного спаривания, приводящие к возникновению неканонических пар и, следовательно, к дефектам в уотсон-криковской спирали. В нормальном цикле репликации такая ошибка может случайно произойти вследствие включения неправильного основания. Спонтанная частота ошибок определяется прежде всего точностью фермента ДНК-полимеразы, отвечающей за репликацию (см. гл. 32). Существует также более ограниченный репара-тивный синтез ДНК, который активируется в результате генетической рекомбинации или повреждения ДНК (см. гл. 34). Различные системы репарации характеризуются разной частотой ошибок. Например, одна из репара-тивных систем Е. соИ особенно часто делает ошибки, и, следовательно, ее активация может стимулировать образование мутаций. Мы не располагаем достаточной информацией о частоте возникновения мутаций такого рода. [c.38]
Что представляет собой механизм появления ошибок Можно предположить, что определенный компонент пути репарации обусловливает продолжение репликации за сайтом повреждения. Когда ДНК-полимераза минует тиминовый димер, она включает неправильные основания и это приводит к появлению мутации. Существуют доказательства того, что для индукции ошибок необходимо присутствие ДНК-полимеразы III, обычной репликазы. Следовательно, рассматриваемая функция действует согласованно с нормальным реплика-ционным аппаратом. Мутации в гене, получившем название итиС, устраняют УФ-индуцируемый мутагенез, но не нарушают какие-либо известные ферментативные функции. Вероятно, продукт этого гена, итиС, служит компонентом системы, продуцирующей ошибки. [c.440]
Модель Уотсона-Крика позволяет представить себе, как может удваиваться нативная молекула ДНК, образуя две одинаковые дочерние молекулы. Поскольку две цепи ДНК комплементарны, каждая из них при расплетании двойной спирали может служить матрицей для синтеза новой комплементарной цепи. Последовательность оснований во вновь синтезируемой цепи будет определяться спецификой водородных связей между основаниями цепи-щаблона и вновь образуемой цепи (рис. 4.13). Таким образом, генетическая информация, содержавшаяся в последовательности пар оснований родительской молекулы, будет полностью воспроизведена в двух дочерних молекулах. Более того, если в процессе удвоения ДНК произошла ошибка и какой-то нуклеотид во вновь образуемой цепи выпал или оказался некомплементарным исходному, то это может изменить информационное содержание молекулы, причем можно ожидать, что эта ошибка будет передана дочерним молекулам ДНК в следующих поколениях. Такая замена пары нуклеотидов может обладать свойствами генетических мутаций. Таким образом, модель структуры ДНК Уотсона и Крика объясняет как способность генов к самоудвоению (репликации), так и их информационные свойства. [c.107]
Несмотря на корректорские функции, присущие ДНК-полимеразам Е. соИ, некоторые нуклеотиды оказываются все же ошибочно включенными в новообразованную цепь ДНК. Их присутствие делает возможным возникновение спонтанных мутаций, в том случае если ошибки не будут исправлены до начала следующего цикла репликации. Свидетельства в пользу существования пострепликационных систем исправления ошибок, или репарации, были получены при изучении таких явлений, как [c.122]
Спонтанные трацзиции могут происходить при репликации ДНК вследствие таутомеризации, т. е. изменения положения протона, меняющего химические свойства молекулы. Таутомеризация в нуклеотидных основаниях меняет их способность образовывать водородные связи, так что аденин приобретает свойства гуанина, гуанин-аденина, цитозин-тимина, а тимин-цитозина (рис. 20.2). Мутагенная активность 5-бром-урацила, аналога тимина, в котором метиловая группа замещена атомом брома, обусловлена таутомеризацией, связанной с большим, нежели у метиловой группы, сродством к электрону атома брома по сравнению с метиловой группой (рис. 20.3). Индуцируемые 5-бромура-цилом мутации могут обусловливаться либо ошибками при включении, [c.9]